물질대사
| 분류:생화학의 일부 |
| 생화학 |
|---|
 |

물질대사(物質代謝, 영어: metabolism), 신진대사 또는 단순히 대사(代謝)는 생물의 세포에서 생명을 유지하기 위해 일어나는 모든 물질의 변화로, 생체내 물질의 분해 · 합성과 같은 모든 화학 작용이다. 효소가 반응을 촉매하며, 대사를 통해 생물은 성장하고 번식하며, 구조를 유지하고 환경에 반응한다. 대사라는 단어는 소화와 세포 간에 물질 수송 등을 포함하여 생물체 내에서 일어나는 모든 화학 반응을 의미하기도 한다. 이 경우 세포 내에서 일어나는 반응의 일부를 중간 대사(영어: intermediary metabolism, intermediate metabolism)라 한다.
대사는 대개 두 부류로 나뉜다. 이화작용(catabolism)은 세포 호흡을 통하여 유기 분자를 분해하고 에너지를 얻는 반응이다. 동화작용(anabolism)은 에너지를 이용하여 단백질이나 핵산과 같은 세포의 구성 성분을 합성하는 반응이다.
대사의 화학 반응은 대사경로를 통해 이루어진다. 대사경로에서 한 화합물이 여러 단계의 반응을 거쳐 다른 화합물로 변화하고, 단계마다 다른 효소가 차례로 반응을 촉매한다. 효소는 에너지를 방출하면서 자발적으로 일어나는 반응을 에너지를 요구하는 반응과 짝지어 생명체가 필요로 하는 반응이 일어나게 한다. 효소는 반응이 좀더 빠르게 일어나도록 하는 촉매 역할을 한다. 또 세포 주위의 환경이나 세포에 오는 신호에 반응하기 위해 대사를 조절하는 역할도 한다.
생물의 대사 시스템에 따라 어떤 물질이 영양소인지 독인지가 정해진다. 예를 들어, 황화 수소는 몇몇 원핵생물에게는 양분이지만, 동물들에게는 독이다.[1] 생물체의 대사 속도는 필요한 음식의 양과 음식을 얻는 방법에 영향을 미친다.
생물 종이 다를지라도 기본적인 대사경로와 그 구성 성분은 매우 유사하다.[2] 시트르산 회로를 구성하는 중간체로 널리 알려진 카복실산들은 단세포 세균인 대장균에서부터 거대한 다세포 생물인 코끼리에 이르기까지 알려진 모든 생물에 존재한다.[3] 대사경로는 진화 초기에 등장했으며, 그 효율성 때문에 계속해서 유지된 것으로 추정된다.[4][5]
주요 생화학 분자[편집]
동물과 식물, 미생물의 구조는 대부분 아미노산, 탄수화물, 지질(종종 지방이라고 불림) 이렇게 세 가지 기본적인 분자로 이루어진다[6]. 생명 유지에 필수적이며, 대사는 이 분자들을 만들어 세포와 조직의 구성 성분으로 쓰거나, 분해하여 에너지로 쓰는 화학 반응이다. 주요 생화학 분자는 결합하여 DNA나 단백질 같은 고분자가 된다.
| 분자 종류 | 단량체 이름 | 중합체 이름 | 중합체 예시 |
|---|---|---|---|
| 아미노산 | 아미노산 | 단백질 (폴리펩타이드라고도 함) | 섬유상 단백질과 구형 단백질 |
| 탄수화물 | 단당류 | 다당류 | 녹말, 글리코젠 및 셀룰로스 |
| 핵산 | 뉴클레오타이드 | 폴리뉴클레오타이드 | DNA 및 RNA |
아미노산과 단백질[편집]
단백질은 아미노산이 펩타이드 결합을 통해 결합한 선형 사슬로 이루어져 있다. 단백질 중 다수는 효소로 대사의 화학 반응을 촉매한다. 기타 단백질은 세포 형태를 유지하는 세포골격과 같이 구조나 기계적 기능을 담당한다.[7] 단백질은 또한 세포 신호, 면역 반응, 세포 부착, 막을 통한 능동수송, 세포 주기에 주요 역할을 한다.[8] 아미노산은 또한 일차적으로 에너지를 공급하는 포도당이 부족하거나 세포가 대사적 스트레스 상태일 때[9] 시트르산 회로에 진입하는 탄소원을 제공하여 세포의 에너지 대사에 관여하기도 한다.[10]
지질[편집]

지질은 생화학 분자들 중 가장 다양한 유형으로 나뉜다. 주요 역할로는 생체막 안팎을 구성하는 것과 에너지원으로서의 역할이 있다.[8] 지질은 소수성이나 양친매성 물질로 정의되지만 벤젠이나 클로로폼과 같은 유기 용매에 녹는다.[11] 지방은 지방산과 글리세롤을 포함하는 큰 화합물 무리이다. 글리세롤이 지방산 세 분자에 결합한 에스터를 트라이글리세라이드라 한다.[12] 트라이글리세라이드 구조에서 조금씩 변형된 화합물이 존재한다. 스핑고지질의 스핑고신, 인지질의 인산염과 같은 친수성 기를 예로 들 수 있다. 그 밖에 스테로이드도 주요 지질 유형으로, 콜레스테롤이 여기에 속한다.[13]
탄수화물[편집]

탄수화물은 하이드록시기가 많은 알데하이드나 케톤으로 사슬이나 고리 형태로 존재할 수 있다. 탄수화물은 생물계에서 가장 풍부한 분자이다. 기능도 다양해서 에너지를 저장하고 수송하는 역할(녹말, 글리코젠), 구조적 역할(식물의 셀룰로스, 동물의 키틴)을 한다.[8] 기본적인 탄수화물 단위를 단당류라고 하며, 갈락토스, 과당, 그리고 가장 중요한 포도당이 있다. 단당류는 무한정에 가까운 다양한 방법으로 서로 결합하여 다당류를 형성할 수 있다.[14]
뉴클레오타이드[편집]
핵산인 DNA와 RNA는 뉴클레오타이드의 중합체이다. 각 뉴클레오타이드는 인산기와 리보스 당, 질소 염기로 이루어진다. 핵산은 유전 정보를 저장하는데 사용되며, 전사와 번역 과정을 통해서 유전 정보를 발현시키는 데에 필수적인 요소이다.[8] 유전 정보는 DNA 수선 기작이 있어 보호되고, DNA 복제를 통해 분자 수를 증가시킨다. 바이러스는 HIV처럼 RNA 유전체를 가지면서 역전사를 통하여 바이러스 RNA 유전체로부터 DNA를 만드는 경우도 있다.[15] 스플라이소좀이나 리보솜과 같은 리보자임에 있는 RNA는 화학 반응을 촉매하는 효소로 작용한다. 각각의 뉴클레오사이드는 핵염기에 리보스 당이 결합해서 만들어진다. 염기는 질소를 포함한 고리 구조로, 퓨린과 피리미딘으로 나뉜다. 뉴클레오타이드는 또한 대사에서 작용기 전이 반응에 참여하는 조효소로 작용하기도 한다.[16]
조효소[편집]

대사는 거대한 화학 반응이지만 작용기의 전이와 관련된 몇 가지 기본적인 반응으로 크게 분류할 수 있다.[17] 공통적인 화학 반응 덕분에 세포는 서로 다른 반응에 참여하는 작용기를 운반하는 대사 중간체의 수를 줄일 수 있다.[16] 조효소는 이렇게 작용기를 운반하는 중간체이다. 작용기 전이 반응은 특정한 한 조효소에 의하여 진행되며, 효소는 그 조효소를 생산하고 소비한다. 따라서 조효소는 계속해서 만들어지고, 사용되며, 재활용된다.[18]
주요 조효소 중 하나가 세포 공동의 에너지 통화라 할 수 있는 아데노신 삼인산(ATP)이다. ATP는 서로 다른 화학 반응 간에 화학 에너지를 전달하는데 사용된다. 세포에는 소량의 ATP만 존재하고, 에너지 요구량에 부합하기 위해 세포는 계속해서 ATP를 빠르게 재생성한다.[18] ATP는 동화작용과 이화작용 사이에서 다리 역할을 한다. 이화작용은 분자를 분해하는 과정이고, 동화작용은 분자를 합성하는 과정이다. 이화작용을 통하여 ATP가 생산되고, 동화작용은 ATP를 소모한다. 그 밖에도 ATP는 인산화 반응에서 인산기를 운반하기도 한다.
비타민은 소량이 필요하지만 세포가 생산할 수 없는 유기 화합물이다. 사람의 영양 측면에서, 비타민은 체내에서 변화한 후 대개 조효소 기능을 한다. 예를 들어 수용성 비타민은 세포에서 이용될 때 인산화되거나 뉴클레오타이드와 결합한다.[19] 비타민 B3(나이아신)의 유도체인 니코틴아마이드 아데닌 다이뉴클레오타이드(NAD+)는 전자수용체 역할을 하는 중요한 조효소이다. 수백 가지 종류의 탈수소효소가 각자의 기질에서 전자를 제거하고 NAD+를 NADH로 환원시킨다. 환원된 조효소 형태인 NADH는 수많은 환원효소의 기질이 되어 환원 반응에 참여한다.[20] NAD+와 NADP+는 세포 내에서 두 가지 연관된 형태인 NADH와 NADPH로 존재한다. NAD+/NADH 형은 이화작용에서, NADP+/NADPH는 동화작용에서 각각 더 중요하게 사용된다.
무기질과 보조 인자[편집]
무기 원소는 대사에서 중요한 역할을 한다. 나트륨과 칼륨은 풍부한 반면, 다른 원소들은 소량 존재하면서 기능한다. 포유류 무게의 99%는 탄소, 질소, 칼슘, 나트륨, 염소, 칼륨, 수소, 인, 산소, 그리고 황이 차지한다.[21] 유기 화합물(단백질, 지질, 탄수화물)이 대부분의 탄소와 질소를 포함하며, 산소와 수소는 물로서 존재한다.[21]
풍부한 무기 원소는 이온성 전해질 역할을 한다. 가장 중요한 이온은 나트륨과 칼륨, 칼슘, 마그네슘, 염화 이온, 인산염과 유기 이온인 탄산수소염이다. 막 안팎의 이온 기울기는 정확하게 조절되어 삼투압과 수소 이온 농도를 유지시킨다.[22] 신경과 근육 조직의 기능에 중요한 역할을 하는 활동전위는 세포외액과 세포기질 사이에서 전해질이 교환되면서 만들어진다.[23] 전해질은 세포막에 있는 이온 통로를 통하여 세포에 드나든다. 예를 들어 근육 수축은 이온 통로와 T-소관(T-tubule)을 통한 칼슘, 나트륨, 칼륨의 이동에 의존한다.[24]

전이 금속은 미량 원소로 존재하며, 아연이나 철은 미량 원소 중에도 비교적 많은 편이다.[25][26] 금속 원소는 단백질의 보조 인자로 쓰이며, 카탈레이스같은 효소와 산소를 운반하는 단백질(예를 들어 헤모글로빈)의 활성에 필수적이다.[27] 금속 보조 인자는 단백질의 특이적인 자리에 결합한다. 효소 보조 인자는 촉매 반응 중에 변형될 수 있지만, 촉매 반응이 끝나면 원래의 상태로 되돌아온다. 금속 미량 영양소는 특이적인 수송체에 의해 생물체 내로 흡수되고 사용하지 않을 때는 페리틴이나 메탈로티오네인(metallothionein)과 같은 저장 단백질과 결합한다.[28][29]
이화작용[편집]
이화작용은 큰 분자를 분해하는 대사 과정으로 음식을 분해하고 산화하는 과정이 포함된다. 이화작용으로 동화작용에 필요한 물질을 공급하고 에너지를 얻는다. 이화작용의 정확한 특성은 생물마다 각기 다르며, 필요에 따라서는 생물을 에너지와 탄소를 얻는 원천에 따라 분류할 수 있다(아래 표 참고). 에너지원으로 유기영양생물(organotroph)은 유기물을, 무기영양생물(lithotroph)은 무기물을, 광영양생물(phototroph)은 태양광을 사용한다. 대사는 서로 다르지만 모두 산화환원 반응에 의존하며, 전자를 유기물, 물, 암모니아, 황화 수소, 철 이온(Fe2+)과 같은 환원된 전자공여체에서 산소, 질산염, 황산염과 같은 전자수용체로 전달한다.[30] 동물의 경우 이 반응은 복잡한 유기물을 이산화 탄소와 물과 같은 간단한 분자로 분해하는 형태이다. 식물이나 남세균 등 광합성 생명체는 전자 전달 반응이 에너지를 방출하지 않고 태양광으로부터 에너지를 흡수해 저장하는 용도로 쓰인다.[31]
| 에너지원 | 태양광 | 광- | 영양생물 | ||
| 이미 존재하는 분자 | 화학- | ||||
| 전자공여체 | 유기 화합물 | 유기- | |||
| 무기 화합물 | 무기- | ||||
| 탄소원 | 유기 화합물 | 종속- | |||
| 무기 화합물 | 독립- | ||||
동물에게 가장 흔한 이화작용은 크게 세 단계로 구분한다. 첫째는 단백질, 다당류, 지질 등의 큰 유기 분자를 세포 바깥에서 보다 작은 분자로 소화하는 것이다. 다음으로 이 작은 분자를 세포 내로 흡수하여 에너지를 일부 방출하는 더욱 작은 분자, 대개는 아세틸 조효소 A(아세틸-CoA)로 전환한다. 마지막으로 아세틸 조효소 A는 시트르산 회로와 전자전달계를 통해 물과 이산화탄소로 산화되고, 이 과정에서 방출되는 에너지의 일부는 조효소 NAD+를 NADH로 환원하면서 에너지를 저장한다.
소화[편집]
녹말, 셀룰로스, 단백질 등의 고분자는 세포 내로 빠르게 흡수될 수 없고, 세포 대사에 이용되기 전에 더 작은 단위로 분해되어야 한다. 일반적인 효소 몇몇이 이러한 중합체를 소화한다. 소화 효소에는 단백질을 아미노산으로 분해하는 단백질 가수분해 효소, 다당류를 단당류 같은 단순한 당으로 분해하는 글리코사이드 가수분해효소(글리코시데이스) 등이 있다.
미생물은 단순히 주변에 소화 효소를 분비하고,[32][33] 동물만이 특수한 세포에서 장으로 효소를 분비한다.[34] 세포 바깥에서 효소가 고분자를 분해하여 만들어낸 아미노산과 당은 능동수송 단백질을 통해 세포 내로 운반된다.[35][36]
유기물에서 얻은 에너지[편집]
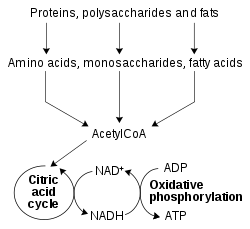
탄수화물 이화작용은 탄수화물을 더 작은 단위로 분해하는 과정이다. 탄수화물은 대개 단당류로 소화된 후에 세포로 유입된다.[37] 세포로 들어온 단당류(포도당이나 과당)는 해당과정을 거치면서 피루브산으로 전환되고 ATP가 만들어진다.[38] 피루브산은 몇몇 대사 회로의 중간체이지만 대부분의 경우 아세틸 조효소 A(아세틸-CoA)로 전환되어 시트르산 회로로 들어간다. 시트르산 회로에서 ATP가 더 많이 생산되기는 하지만, 이 회로에서 가장 중요한 생산물은 아세틸-CoA가 산화되면서 NAD+에서 생산된 NADH이다. 무산소 조건에서는 젖산 탈수소효소의 작용으로 해당과정을 통해 젖산이 생산되고, NADH는 해당과정에서 재사용되기 위해서 NAD+로 재산화된다. 포도당은 오탄당 인산 경로라는 다른 경로로 분해될 수도 있다. 오탄당 인산 경로는 조효소 NADPH를 환원하고, 핵산의 구성 성분인 리보스와 같은 오탄당을 생산하는 과정이다.
지방은 지방산과 글리세롤로 가수분해된다. 글리세롤은 해당과정으로 들어가고 지방산은 베타 산화 과정을 통하여 분해되어 아세틸-CoA를 방출하고, 시트르산 회로에 진입한다. 지방산이 산화되면 산소를 더 많이 가지고 있는 탄수화물보다 더 많은 에너지를 방출할 수 있다. 스테로이드 또한 베타 산화와 유사한 과정을 통해 몇몇 세균에 의해 분해되어 상당한 양의 아세틸-CoA, 프로피오닐-CoA, 피루브산으로 전환되고, 이들은 모두 세포가 에너지를 얻는 데에 쓸 수 있다. 결핵균(M. tuberculosis)은 탄소원으로 콜레스테롤만을 이용하여 증식할 수 있으며, 콜레스테롤 사용 경로와 관련된 유전자가 결핵균이 감염되는 생활사의 여러 단계에서 중요한 역할을 한다는 것이 확인되었다.[39]
아미노산은 단백질이나 다른 생체분자를 합성하는데 이용되거나, 에너지원으로 사용되어 요소와 이산화 탄소로 산화된다.[40] 아미노산의 산화 경로는 아미노기 전이효소에 의해 아미노기를 제거하면서 시작한다. 아미노기는 요소 회로로 들어가고, 아미노기가 제거된 탄소 골격은 케토산의 형태로 남는다. 이 케토산 중에 몇몇은 시트르산 회로의 중간체로, 예를 들어 글루탐산이 탈아미노화되면 α-케토글루타르산이 된다.[41] 어떤 아미노산은 포도당신생합성 과정을 통해 포도당으로 전환될 수 있는데, 이를 당생성 아미노산이라 한다.[42]
에너지 전환[편집]
산화적 인산화[편집]
산화적 인산화 과정에서 전자는 유기 분자에서 제거되어 산소로 옮겨지고 방출된 에너지는 ATP를 만드는데 쓰인다. 이 과정은 진핵생물의 경우 미토콘드리아 막에 있는 단백질 복합체들, 즉 전자전달계에서 일어난다. 원핵생물에서는 세포의 내막에 있는 단백질들에서 일어난다.[43] 이 단백질 복합체들은 환원된 분자(즉, NADH)에서 산소로 전자가 전달되면서 방출되는 에너지를 이용하여 막 바깥으로 양성자를 퍼낸다(pump).[44]

미토콘드리아 기질에서 막 사이 공간으로 양성자를 퍼내면 미토콘드리아 내막을 경계로 안팎으로 양성자의 농도 차가 생겨서 전기화학적 기울기를 형성한다.[45] 이 힘에 의하여 양성자는 ATP 합성효소를 통하여 다시 미토콘드리아로 돌아간다. 양성자 흐름에 따라 ATP 합성효소의 기둥 소단위체(γ 소단위체)가 회전하고, ATP 합성효소 활성 자리의 구조가 변화하여 ADP을 ATP으로 인산화한다.[18]
무기 화합물로부터의 에너지[편집]
화학무기영양생물(chemolithotroph)은 원핵생물의 일종으로 무기 화합물을 산화하여 에너지를 얻는다. 여기 속하는 생물은 수소,[46] 환원형 황 화합물(황화물, 황화 수소, 티오황산염),[1] 철 이온(Fe II),[47] 암모니아[48] 등의 환원력을 이용할 수 있으며, 이들 화합물을 산화하고 산소나 아질산염을 전자수용체로 이용하여 에너지를 얻는다.[49] 무기 화합물에서 에너지를 얻는 미생물의 대사는 이산화 탄소에서 아세트산을 생산하거나(acetogenesis), 질화 작용(nitrification), 탈질소 작용(denitrification) 등 전세계적인 생물지구화학적 순환에 중요한 역할을 하며, 토양비옥도에 필수적인 요소이다.[50][51]
빛에서 얻는 에너지[편집]
식물, 남세균, 녹색유황세균류와 몇몇 원생생물은 태양광에서 에너지를 얻는다. 이 과정은 종종 광합성의 일부로서 이산화 탄소를 유기 화합물로 전환하는 과정을 동반한다. 자색 세균과 녹색황세균이 태양광을 에너지원으로 쓸 수 있듯이 원핵생물은 에너지 포착과 탄소 고정을 각각 따로 시행할 수 있으며, 동시에 대사 방향을 탄소 고정이나 유기 화합물의 발효로 전환할 수 있다.[52][53]
태양광을 에너지로 저장하는 생물 역시 양성자 농도 기울기를 이용하여 에너지를 저장하므로 원리상 산화적 인산화와 유사하다. 양성자 농도 기울기에 의한 힘이 ATP 합성을 유도한다.[18] 전자전달계를 돌리기 위한 전자는 광합성 반응 중심 또는 로돕신이라는 빛을 모으는 단백질로부터 온다. 반응 중심은 그곳에 존재하는 광합성 색소의 종류에 따라 두 부류로 나뉜다. 광합성 세균은 대개 한 종류의 반응 중심을 가지고, 식물과 남세균은 두 종류를 모두 가진다.[54]
식물과 조류, 남세균은 광계 II에서 빛 에너지를 받아 물에서 전자를 제거하고 부산물로 산소를 방출한다. 전자는 사이토크롬 b6f 복합체로 전달되고, 그 에너지로 엽록체의 틸라코이드 막 안쪽으로 양성자를 퍼낸다.[8] 양성자는 다시 막을 가로질러 확산되면서 ATP 합성효소를 작동시킨다. 전자는 광계 I으로 전달되어 조효소 NADP+를 환원시켜 NADPH가 캘빈 회로에서 사용되게 하거나, ATP 생성을 계속하기 위해 재활용된다.[55]
동화작용[편집]
동화작용은 이화작용에서 방출된 에너지로 복잡한 분자를 합성하는데 이용하는 대사 과정이다. 일반적으로 세포의 구조를 이루는 복잡한 분자는 작고 단순한 전구물질로부터 차례로 합성된다. 동화작용은 기본적으로 세 단계를 거친다. 처음에는 아미노산, 단당류, 아이소프레노이드, 뉴클레오타이드와 같은 전구물질을 생산하고, 그 다음에는 ATP에 저장된 에너지를 이용하여 전구물질을 활성이 있는 형태로 바꾼다. 마지막으로 이 전구물질들을 조립하여 단백질, 다당류, 지질, 핵산 등의 복잡한 분자들을 생산한다.
생물에 따라 스스로 세포 내에서 만들 수 있는 분자들의 종류가 각기 다르다. 식물과 같은 자가영양생물은 이산화 탄소나 물처럼 간단한 분자에서 다당류나 단백질을 합성할 수 있다. 반면에 종속영양생물은 보다 복잡한 물질을 합성하기 위한 전구물질들, 예컨대 단당류나 아미노산을 필요로 한다. 생물은 궁극적인 에너지원에 따라 더 세분화된다. 광합성독립영양생물(photoautotroph)과 광합성종속영양생물(photoheterotroph)은 빛에서 에너지를 얻고, 화학독립영양생물(chemoautotroph)과 화학종속영양생물(chemoheterotroph)은 무기 산화 반응을 통하여 에너지를 얻는다.
탄소 고정[편집]

광합성은 햇빛과 이산화 탄소(CO2)를 이용하여 탄수화물을 합성하는 과정이다. 식물과 남세균, 조류는 광합성을 통해 물을 분해하고 산소를 부산물로 생산한다. 이 과정은 광합성 반응 중심에서 생산된 ATP와 NADPH를 사용하여 CO2를 3-포스포글리세르산으로 전환하고, 3-포스포글리세르산은 이후 포도당으로 전환된다. 탄소 고정 반응은 캘빈 회로의 일부로 루비스코(RuBisCO)라는 효소가 참여한다.[56] 식물에서는 C3 탄소 고정, C4 탄소 고정, 그리고 CAM 광합성 이렇게 세 종류의 광합성이 일어나며, 이들은 CO2가 캘빈 회로에 들어가는 방법에 따라 구분된다. C3 식물은 CO2를 직접적으로 고정하고, C4 식물과 CAM 식물은 CO2를 먼저 다른 화합물의 일부로 포함시킨다. 이는 강렬한 햇빛과 건조한 환경에 적응하기 위한 방편이다.[57]
광합성 원핵생물의 경우 탄소 고정 기작이 좀 더 다양하다. 캘빈 회로, 역방향 시트르산 회로(reversed citric acid cycle),[58] 혹은 아세틸-CoA의 카복실화를 통해 이산화 탄소를 고정한다.[59][60] 화학독립영양(chemoautotroph) 원핵생물 또한 캘빈 회로를 통해 이산화 탄소를 고정하지만, 반응을 진행하기 위한 에너지는 무기 화합물에서 얻는다.[61]
탄수화물과 글리칸[편집]
단순한 유기산은 포도당과 같은 단당류로 전환될 수 있고, 그 후에 녹말 같은 다당류로 합성된다. 피루브산, 젖산, 글리세롤, 3-포스포글리세르산 및 아미노산 등에서 포도당을 생성하는 것을 포도당신생합성이라고 한다. 포도당신생합성은 해당과정과 많은 부분에서 겹치는 중간체들을 통해 피루브산을 포도당 6-인산으로 전환한다.[38] 그러나 포도당신생합성 경로에 참여하는 효소들 중에는 해당과정에 참여하지 않는 효소가 있으므로 포도당신생합성은 단순히 해당과정의 역반응은 아니다. 이로 인하여 포도당을 생성하고 분해하는 것이 각기 따로 조절될 수 있으며, 두 대사 경로가 에너지만 소모하는 무익 회로(futile cycle)로 동시에 작동하는 것을 방지한다.[62][63]
에너지를 저장하는 일반적인 분자는 지방이지만, 척추동물의 경우 지방산은 포도당신생합성을 통하여 포도당으로 전환될 수 없다. 이는 식물과 달리 동물에게는 아세틸-CoA를 피루브산으로 전환하는데 필요한 효소 기구가 없기 때문이다.[64] 따라서 장기간 기아 상태에서 척추동물은 지방산을 대사하지 못하는 조직(예를 들어 뇌)이 포도당 대신에 사용할 케톤체를 생산해야 한다.[65] 식물과 세균 등 다른 생물은 시트르산 회로에서 탈카복실화 반응을 우회하여, 아세틸-CoA를 옥살아세트산으로 변환하고 포도당 생산에 이용될 수 있게 하는 글리옥실산 회로를 이용하여 이 문제를 해결한다.[64][66]
다당류와 글리칸은 유리딘 이인산 글루코스(UDP-glucose)와 같은 반응성 당인산을 중합되고 있는 다당류의 하이드록시기에 순차적으로 부가하는 글리코실전이효소(glycosyltransferase)에 의해 생성된다. 기질 고리 구조에 있는 하이드록시기라면 당이 중합될 수 있기 때문에 다당류는 사슬 구조와 가지친 형태 모두가 될 수 있다.[67] 생성된 다당류는 그 자체로 구조적 기능이나 대사적 기능을 하거나, 올리고당전이효소(oligosaccharyltransferase)에 의해 지질이나 단백질에 결합할 수도 있다.[68][69]
지방산, 아이소프레노이드 및 스테로이드[편집]

지방산은 아세틸-CoA 단위체를 중합하고 환원하는 지방산 생성효소(fatty acid synthase)에 의해 만들어진다. 지방산의 아실 사슬은 아실기를 더하고 알코올로 환원한 후, 알켄으로 탈수소화하고 다시 알케인으로 환원하는 일련의 반응에 의해 신장된다. 지방산 생합성에 관여하는 효소는 두 그룹으로 나뉜다. 동물과 균류는 여러 기능을 가진 제1형 단백질 하나에 의해 지방산을 합성하고,[70] 식물의 색소체와 세균은 제2형 단백질이 합성 경로의 각 단계에 관여한다.[71][72]
테르펜과 아이소프레노이드는 카로티노이드를 포함하는 지질의 큰 부류로 식물에서 생산되는 천연물 중 가장 큰 집단이다.[73] 이 화합물은 반응성 전구체인 아이소펜테닐 피로인산과 다이메틸알릴 피로인산에서 받은 아이소프렌 단위체의 중합과 변형을 통해 생산된다.[74] 이러한 전구체들은 서로 다른 경로로 합성된다. 동물과 고균은 아세틸-CoA에서 시작해서 메발론산 경로를 통해 전구체를 합성하고,[75] 식물과 세균은 피루브산과 글리세르알데하이드 3-인산을 이용하여 합성한다.[74][76] 활성화된 아이소프렌 공여체를 이용하는 주요 반응 중 하나가 스테로이드 생합성 경로이다. 스테로이드 합성 경로에서 아이소프렌 단위체는 결합하여 스쿠알렌이 되고, 고리를 형성하여 라노스테롤이 된다.[77] 라노스테롤은 콜레스테롤과 에르고스테롤 등 다른 스테로이드로 전환된다.[77][78]
단백질[편집]
생물에 따라 20가지의 아미노산을 합성할 수 있는 능력에 차이가 있다. 대부분의 세균과 식물은 20가지 전부를 합성할 수 있지만, 포유류는 오직 열한 가지의 비필수 아미노산만을 합성할 수 있고 나머지 아홉 가지의 필수 아미노산은 음식물에서 얻어야 한다.[8] 마이코플라스마(Mycoplasma pneumoniae)과 같이 단순한 기생충은 모든 아미노산을 합성할 수 없고 숙주로부터 직접 아미노산을 탈취한다.[79] 모든 아미노산은 해당과정, 시트르산 회로, 혹은 오탄당 인산 경로의 중간생성물로부터 합성된다. 질소는 글루탐산과 글루타민에서 공급된다. 아미노산은 적절한 α-케토산의 형성에 의존하여 합성되고, α-케토산은 아미노기 전달반응에 의하여 아미노산이 된다.[80]
아미노산은 펩타이드 결합에 의해 사슬형으로 연결되고 단백질이 된다. 각각의 단백질은 특이적인 아미노산 서열로 이루어지며, 이를 일차 구조라 한다. 아미노산은 다양한 서열로 결합하여 셀 수 없이 다양한 단백질을 형성한다. 단백질이 형성될 때 아미노산은 에스터 결합으로 운반 RNA(tRNA)에 붙어서 활성화된다. 이 아미노아실-tRNA 전구체는 ATP를 이용하는 아미노아실 tRNA 합성효소에 의해 생산된다.[81] 아미노아실-tRNA는 리보솜의 기질이 되고, 리보솜은 전령 RNA에 있는 서열 정보에 따라 아미노산을 신장하는 단백질 사슬에 결합시킨다.[82]
뉴클레오타이드 합성과 회수[편집]
뉴클레오타이드는 아미노산, 이산화 탄소, 폼산으로부터 만들어지는데 이러한 대사에는 막대한 에너지가 소모된다.[83] 따라서 대부분의 생물들은 이미 만들어져 있는 뉴클레오타이드를 효과적으로 회수하는 체계를 발달시켰다.[83][84] 퓨린은 뉴클레오사이드(리보스에 결합한 염기) 형태로 합성된다.[85] 아데닌과 구아닌은 뉴클레오사이드 전구체인 이노신 일인산에서 합성되며, 아미노산 글라이신, 글루타민, 아스파르트산 및 조효소 테트라하이드로엽산에서 전이된 폼산에서 원자를 이용한다. 피리미딘은 글루타민과 아스파르트산에서 형성된 오로트산으로부터 합성된다.[86]
생체이물 및 산화환원 대사[편집]
모든 생물은 영양분으로 사용할 수 없는 화합물에 지속적으로 노출되고, 대사적 기능이 없는 이물질이 세포 내에 축적되면 해로울 수 있다. 이렇게 유해성을 가진 물질을 생체이물이라 한다.[87] 합성 약물, 자연 독소, 항생제 같은 생체이물은 생체이물을 대사하는 효소에 의하여 해독된다. 사람의 해독 효소에는 사이토크롬 P450 산화효소,[88] UDP-글루쿠로노실전이효소(glucuronosyltransferase),[89] 글루타티온 S-전이효소 등이 있다.[90] 이 효소계는 생체이물을 산화하고(1기), 생체이물 분자에 수용성 작용기를 부가한 후(2기), 세포 밖으로 퍼내거나 다세포 생물의 경우 계속해서 대사 과정을 거쳐 배출시킨다(3기). 생태학에서 이 반응은 특히 미생물의 오염 물질 생분해와 오염된 땅과 기름 유출에 대한 생물적 환경정화에 중요한 역할을 한다.[91] 미생물의 반응은 많은 부분 다세포 생물의 반응과 유사하지만, 미생물은 특유의 엄청난 다양성으로 다세포 생물에 비하여 훨씬 많은 종류의 생체이물을 처리하며, 유기염소 화합물과 같이 잔류성 유기 오염 물질까지도 처리할 수 있다.[92]
한편 호기성 생물에게는 산화적 스트레스 문제가 있다.[93] 산화적 인산화와 이황 결합 등의 과정은 과산화수소 같은 활성산소를 생성한다.[94] 유해 산화제는 글루타티온 같은 항산화 대사체나 카탈레이스 혹은 과산화효소 등에 의해 제거된다.[95][96]
생물의 열역학[편집]
열과 일의 전달에 대한 열역학 법칙은 생물에게도 예외가 아니다. 열역학 제2법칙에 따르면 어떤 닫힌계에서 전체 엔트로피(무질서도)의 총량은 증가하는 경향이 있다. 생물은 놀라운 복잡성으로 이 법칙에 모순되는 것 같지만, 생물은 주변 환경과 물질과 에너지를 교환하는 열린계로 존재한다. 따라서 살아있는 계는 평형 상태가 아니며, 환경의 엔트로피를 높이면서 고도의 복잡성을 유지하는 흩어지기계(dissipative system)이다.[97] 세포는 이화작용의 자발적 과정과 동화작용의 비자발적 과정을 짝지어 대사가 일어나도록 한다. 열역학적 용어로, 대사는 무질서를 창조하면서 질서를 유지한다.[98]
조절과 통제[편집]
생물이 있는 환경은 계속해서 변하기 때문에 대사는 세포 내의 일정한 조건, 즉 항상성을 유지하기 위해 섬세하게 조절되어야 한다.[99][100] 대사 조절은 또한 생물이 신호에 반응하고 환경과 적절히 상호작용할 수 있도록 한다.[101] 어떻게 대사 경로가 통제되는지 이해하는데에는 아래의 밀접하게 연결된 두 개념이 중요한 역할을 한다.
- 대사 경로에서 효소를 조절(regulation)한다는 것은 어떻게 효소가 신호에 반응하여 활성이 증가하고 감소하는지를 의미한다.
- 효소가 발휘하는 통제(control)란 활성이 변화한 효소가 전체 대사 경로의 속도(혹은 흐름)에 미치는 영향을 의미한다.[102]
예를 들어 어떤 효소의 활성이 크게 변화하더라도(매우 잘 조절됨) 이 변화로 인하여 대사 경로의 흐름에 큰 변화가 없다면, 이 효소는 해당 대사 경로의 통제에 관여하지 않는다.[103]

대사 조절에는 다양한 단계가 있다. 내인성 조절은 대사 회로가 기질이나 산물의 수준에 따라 스스로를 조절하는 것이다. 예를 들어 산물이 양이 감소하면 그것을 보상하기 위해 그 경로로의 흐름이 증가한다.[102] 이런 경우에는 종종 다른 자리 입체성 조절로 효소의 활성을 조절한다.[104] 외인성 통제는 다세포 생물의 세포가 다른 세포로부터 온 신호에 반응하여 대사를 변경할 때 나타난다. 이 때 신호는 대개 호르몬이나 성장 인자 형태로, 세포 표면에 있는 특수한 수용체에 의해 감지된다.[105] 신호는 단백질 인산화 연쇄 반응을 거쳐 이차 신호 전달계에 의해 세포 내로 전달된다.[106]
외인성 조절의 예로 잘 알려진 것이 인슐린 호르몬에 의한 포도당 대사의 조절이다.[107] 인슐린은 혈당량의 증가에 따라 생산된다. 인슐린이 수용체에 결합하면 단백질 인산화효소 연쇄반응을 활성화하여 세포로 하여금 포도당을 흡수하여 지방산이나 글리코겐 등 저장형 분자로 전환하게 한다.[108] 포도당의 대사는 글리코겐을 분해하는 인산가수분해효소와 합성하는 글리코겐 합성효소의 활성에 따라 통제된다. 이 두 효소는 인산화를 통해 상호조절된다. 즉, 글리코겐 분해효소는 인산화되면 활성화되고, 합성효소는 저해된다. 인슐린은 단백질 인산분해효소를 활성화하고, 따라서 글리코겐 분해 및 합성효소의 인산화 정도를 감소시켜 글리코겐이 합성되도록 유도한다.[109]
진화[편집]

위에서 기술한 해당과정, 시트르산 회로와 같은 주요 대사 경로들은 모든 생물 도메인에 존재하며 모든 생물의 공통 조상에도 있었다.[3][110] 공통 조상 세포는 원핵세포였으며 아마도 광범위한 아미노산, 뉴클레오타이드, 탄수화물, 지질 대사를 갖춘 메테인 세균이었을 것이다.[111][112] 진화를 거치면서 오래된 대사 경로를 계속 유지한 것은 그 대사가 특정한 대사적 난제를 해결하는 최적의 방법이기 때문일 것이다. 해당과정과 시트르산 회로의 경우 최소의 단계로 매우 효율적인 최종 산물을 생산한다.[4][5] 비부호 DNA 단편에 영향을 미치는 돌연변이는 대사 효율에만 영향을 미칠 것이다.[113] 효소를 이용한 대사 중 가장 먼저 등장한 것은 퓨린 뉴클레오타이드 대사의 일부이고, 이전의 대사 경로는 고대 RNA 세계의 일부였을 것이다.[114]
새로운 대사 경로가 진화한 기작을 설명하기 위한 모델이 여럿 존재한다. 원형의 대사 경로에 이전부터 존재하던 효소가 새로운 반응 경로에 통합될 뿐만 아니라 새로운 효소가 차례로 더해지고 복제되어 전체 경로가 분지하였다는 모델이 있다.[115] 각 기작의 상대적인 중요도는 정확하지 않으나, 유전체 연구를 통하여 대사 경로의 효소가 공동의 조상을 가진다는 것이 밝혀졌으며 따라서 이미 존재하던 경로에서 차례로 새로운 기능을 가진 경로로 진화했을 것으로 추정하고 있다.[116][117] 대사 네트워크의 단백질 구조의 진화를 추적하는 연구에서 나온 대안적인 모델에 따르면, 효소는 서로 다른 대사 경로에서 유사한 기능을 수행하기 위해 널리 동원되었을 것이다.[118] 그 결과 효소의 진화적 모자이크가 나타나는 것이다.[119] 세 번째 가능성은 대사의 일정 부분이 특정 단위(module)로서 서로 다른 경로에서 재활용되고 여러 분자에 대해 유사한 기능을 한다는 것이다.[120]
진화를 통해 새로운 대사 경로가 생겨나는 것과 마찬가지로 대사 기능을 잃을 수 있다. 예를 들어 몇몇 기생충은 생존에 필수적이지 않은 대사 과정이 퇴화하고 숙주로부터 아미노산, 뉴클레오타이드, 탄수화물을 얻게 되었다.[121][122] 세포내 공생하는 생물에서 이와 유사한 대사 능력 감소를 찾아볼 수 있다.[123]
연구 및 조작[편집]

전통적으로 물질대사는 환원주의적 관점에서 단일 대사 경로에 초점을 맞춘 연구 대상이었다. 특히 귀중한 연구 도구인 방사성 추적자를 사용하여 전구물질에서 최종 산물까지 경로를 생물 전체나 조직, 세포 수준에서 규명할 수 있다.[124] 화학 반응을 촉진하는 효소를 정제하여 저해제에 대한 반응과 속도를 조사한다. 동시에 세포나 조직 안에 있는 작은 분자를 밝혀내고, 이 분자들의 총체적 집합을 대사체라 한다. 이러한 연구를 통하여 단순한 대사 경로의 구조와 기능에 대한 이해를 높일 수 있지만, 완전한 세포의 대사와 같은 더 복잡한 계에 적용하기에는 무리가 있다.[125]
수천 가지의 효소가 공존하는 세포 내 대사 네트워크의 복잡성이란 것이 어떤 개념인지는 불과 43개의 단백질과 40개의 대사산물 사이의 상호작용을 나타낸 오른쪽의 그림에서 볼 수 있다. 유전체 서열에는 최대 45,000개에 이르는 유전자 목록이 있다.[126] 이제는 유전체 자료를 이용하여 전체 생화학 반응의 네트워크를 재구성하고, 그 행동을 설명하고 예측하는 전체적인 수학적 모델을 산출하는 것이 가능하다.[127] 특히 전통적인 방법으로 얻은 대사 경로와 대사산물 자료를 단백체학과 DNA 마이크로어레이 연구에서 얻은 유전자 발현 자료에 통합할 때 수학적 모델이 유용하다.[128] 이러한 기술을 통하여 사람의 대사에 대한 모델이 정립되고, 의약품 개발이나 생화학 연구에 유용하게 쓰일 수 있게 되었다.[129] 사람의 대사 모델은 같은 단백질이나 대사산물을 공유하는 질병을 분류하기 위한 네트워크 분석에 이용되고 있다.[130][131]
세균의 대사 네트워크는 나비 넥타이[132][133][134] 조직의 좋은 예이다. 나비 넥타이 구조는 광범위한 영양물질을 받아들여 상대적으로 적은 중간체를 통해 다양한 산물과 복잡한 고분자를 생산할 수 있는 구조이다.
대사 공학은 주요한 대사 정보 응용 기술이다. 효모, 식물 및 세균은 생명공학기술에 유용하게 유전적으로 변형되어 항생제나 의약품 혹은 1,3-프로판디올(1,3-propanediol), 시킴산 같은 산업용 화합물 생산에 이용된다.[135][136][137] 유전자 변형은 대개 생산하는데 드는 에너지 양을 줄이고, 생산량을 증대시키며, 폐기물을 감소시키는 것을 목적으로 한다.[138]
역사[편집]

물질대사라는 용어는 변화 혹은 전복(顚覆, overthrow)을 뜻하는 그리스어: Μεταβολισμός 에서 유래하였다.[139] 1260년 이븐 알나피스(Ibn al-Nafis는 저서에서 "인체와 각 부분은 지속적인 소멸과 생성 상태에 있으며, 따라서 불가피하게 영구적인 변화를 겪는다"고 서술하면서 물질대사의 개념을 처음으로 문서화하였다.[140] 물질대사에 대한 과학적 연구의 역사는 몇 세기에 걸쳐 나타난다. 동물 전체를 연구하던 초기 시기에서 개인의 대사 반응을 연구하는 현대의 생화학으로 발전하였다. 사람의 대사에 대한 첫 통제 실험은 1614년 출판된 산토리오 산토리오의 저서 Ars de statica medicina에서 나타난다.[141] 그는 일상에서 각종 활동을 하는 동안 스스로 무게 변화를 측정하고, 섭취한 음식 무게의 대부분은 잃는다는 것을 발견하였다. 그는 이것을 "인식할 수 없는 호흡"이라 불렀다.
초기에 물질대사 기작은 밝혀지지 않았고 어떤 힘이 살아있는 조직에 생기를 불어넣는다고 생각하였다.[142] 19세기에 효모에 의하여 설탕이 알코올로 발효되는 것을 관찰한 루이 파스퇴르는 발효가 효모 세포 안에 있는 물질이 발효를 촉매 작용한다고 결론지었다. 그는 "알코올성 발효는 효모 세포의 죽음이나 부패가 아니라 삶과 연관된 활동이다"고 기술하였다.[143] 이 발견과 1828년 프리드리히 뵐러의 요소의 화학적 합성에 대한 논문[144]은 전적으로 무기 전구체만으로 유기 화합물 합성을 논한 중요한 발견이다. 이로써 세포에서 발견된 유기 화합물이나 화학 반응이 일반적인 화학 반응의 원리와 다를 것이 없음이 증명되었다.
에두아르트 부흐너가 20세기 초 효소를 발견함에 따라 세포의 생물학적 연구에서 물질대사의 화학 반응을 분리하여 연구할 수 있게 되었으며, 이것이 생화학의 시초가 되었다.[145] 생화학의 지식은 20세기 초에 급격하게 증가하였다. 가장 많은 연구를 한 근대 생화학자 중 한 명은 크레브스(독일어: Hans Adolf Krebs)로, 물질대사 연구에 큰 기여를 하였다.[146] 그는 요소 회로를 발견하였고, 후에 한스 콘버그(Hans Kornberg)와 함께 시트르산 회로와 글리옥실산 회로를 발견하였다.[147][148][66] 현대 생화학은 크로마토그래피, X선 회절을 이용한 분광학, NMR 분광법, 방사성 동위원소 표지, 전자 현미경, 분자 역학 시뮬레이션 등 새로운 기술의 발달에 힘입어 크게 발전하였다. 이러한 기술들을 통해 세포에 있는 분자 및 물질대사 경로들을 발견하고 상세히 분석할 수 있게 되었다.
같이 보기[편집]
각주[편집]
- ↑ 가 나 Friedrich C (1998). “Physiology and genetics of sulfur-oxidizing bacteria”. 《Adv Microb Physiol》. Advances in Microbial Physiology 39: 235–89. doi:10.1016/S0065-2911(08)60018-1. ISBN 9780120277391. PMID 9328649.
- ↑ Pace NR (January 2001). “The universal nature of biochemistry”. 《Proc. Natl. Acad. Sci. U.S.A.》 98 (3): 805–8. Bibcode:2001PNAS...98..805P. doi:10.1073/pnas.98.3.805. PMC 33372. PMID 11158550.
- ↑ 가 나 Smith E, Morowitz H (2004). “Universality in intermediary metabolism”. 《Proc Natl Acad Sci USA》 101 (36): 13168–73. Bibcode:2004PNAS..10113168S. doi:10.1073/pnas.0404922101. PMC 516543. PMID 15340153.
- ↑ 가 나 Ebenhöh O, Heinrich R (2001). “Evolutionary optimization of metabolic pathways. Theoretical reconstruction of the stoichiometry of ATP and NADH producing systems”. 《Bull Math Biol》 63 (1): 21–55. doi:10.1006/bulm.2000.0197. PMID 11146883.
- ↑ 가 나 Meléndez-Hevia E, Waddell T, Cascante M (1996). “The puzzle of the Krebs citric acid cycle: assembling the pieces of chemically feasible reactions, and opportunism in the design of metabolic pathways during evolution”. 《J Mol Evol》 43 (3): 293–303. doi:10.1007/BF02338838. PMID 8703096.
- ↑ Pfeiffer, AF (2013). “Fettreiche Ernährung bei Diabetes mellitus”. 《Dtsch Med Wochenschr》 138 (18): 964–966. doi:10.1055/s-0032-1333058.
- ↑ Michie K, Löwe J (2006). “Dynamic filaments of the bacterial cytoskeleton”. 《Annu Rev Biochem》 75: 467–92. doi:10.1146/annurev.biochem.75.103004.142452. PMID 16756499.
- ↑ 가 나 다 라 마 바 Nelson, David L.; Michael M. Cox (2005). 《Lehninger Principles of Biochemistry》. New York: W. H. Freeman and company. 841쪽. ISBN 0-7167-4339-6.
- ↑ Hothersall, J and Ahmed, A (2013). “Metabolic fate of the increased yeast amino acid uptake subsequent to catabolite derepression”. 《J Amino Acids》 2013: e461901. doi:10.1155/2013/461901. PMC 3575661. PMID 23431419.
- ↑ Kelleher, J,Bryan 3rd, B, Mallet,R, Holleran, A, Murphy, A, and Fiskum, G (1987). “Analysis of tricarboxylic acid-cycle metabolism of hepatoma cells by comparison of 14CO2 ratios”. 《Biochem J》 246 (3): 633–639. PMC 346906. PMID 6752947.
- ↑ Fahy E, Subramaniam S, Brown H, Glass C, Merrill A, Murphy R, Raetz C, Russell D, Seyama Y, Shaw W, Shimizu T, Spener F, van Meer G, VanNieuwenhze M, White S, Witztum J, Dennis E (2005). “A comprehensive classification system for lipids”. 《J Lipid Res》 46 (5): 839–61. doi:10.1194/jlr.E400004-JLR200. PMID 15722563. 2010년 8월 24일에 원본 문서에서 보존된 문서. 2014년 10월 7일에 확인함.
- ↑ “Nomenclature of Lipids”. IUPAC-IUB Commission on Biochemical Nomenclature (CBN). 2007년 3월 8일에 확인함.
- ↑ Hegardt F (1999). “Mitochondrial 3-hydroxy-3-methylglutaryl-CoA synthase: a control enzyme in ketogenesis”. 《Biochem J》 338 (Pt 3): 569–82. doi:10.1042/0264-6021:3380569. PMC 1220089. PMID 10051425.
- ↑ Raman R, Raguram S, Venkataraman G, Paulson J, Sasisekharan R (2005). “Glycomics: an integrated systems approach to structure-function relationships of glycans”. 《Nat Methods》 2 (11): 817–24. doi:10.1038/nmeth807. PMID 16278650.
- ↑ Sierra S, Kupfer B, Kaiser R (2005). “Basics of the virology of HIV-1 and its replication”. 《J Clin Virol》 34 (4): 233–44. doi:10.1016/j.jcv.2005.09.004. PMID 16198625.
- ↑ 가 나 Wimmer M, Rose I (1978). “Mechanisms of enzyme-catalyzed group transfer reactions”. 《Annu Rev Biochem》 47: 1031–78. doi:10.1146/annurev.bi.47.070178.005123. PMID 354490.
- ↑ Mitchell P (1979). “The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems”. 《Eur J Biochem》 95 (1): 1–20. doi:10.1111/j.1432-1033.1979.tb12934.x. PMID 378655.
- ↑ 가 나 다 라 Dimroth P, von Ballmoos C, Meier T (March 2006). “Catalytic and mechanical cycles in F-ATP synthases: Fourth in the Cycles Review Series”. 《EMBO Rep》 7 (3): 276–82. doi:10.1038/sj.embor.7400646. PMC 1456893. PMID 16607397.
- ↑ Coulston, Ann; Kerner, John; Hattner, JoAnn; Srivastava, Ashini (2006). 〈Nutrition Principles and Clinical Nutrition〉. 《Stanford School of Medicine Nutrition Courses》. SUMMIT.
- ↑ Pollak N, Dölle C, Ziegler M (2007). “The power to reduce: pyridine nucleotides – small molecules with a multitude of functions”. 《Biochem J》 402 (2): 205–18. doi:10.1042/BJ20061638. PMC 1798440. PMID 17295611.
- ↑ 가 나 Heymsfield S, Waki M, Kehayias J, Lichtman S, Dilmanian F, Kamen Y, Wang J, Pierson R (1991). “Chemical and elemental analysis of humans in vivo using improved body composition models”. 《Am J Physiol》 261 (2 Pt 1): E190–8. PMID 1872381.
- ↑ Sychrová H (2004). “Yeast as a model organism to study transport and homeostasis of alkali metal cations” (PDF). 《Physiol Res》. 53 Suppl 1: S91–8. PMID 15119939.
- ↑ Levitan I (1988). “Modulation of ion channels in neurons and other cells”. 《Annu Rev Neurosci》 11: 119–36. doi:10.1146/annurev.ne.11.030188.001003. PMID 2452594.
- ↑ Dulhunty A (2006). “Excitation-contraction coupling from the 1950s into the new millennium”. 《Clin Exp Pharmacol Physiol》 33 (9): 763–72. doi:10.1111/j.1440-1681.2006.04441.x. PMID 16922804.
- ↑ Mahan D, Shields R (1998). “Macro- and micromineral composition of pigs from birth to 145 kilograms of body weight”. 《J Anim Sci》 76 (2): 506–12. PMID 9498359. 2011년 4월 30일에 원본 문서에서 보존된 문서. 2011년 4월 30일에 확인함.
- ↑ Husted S, Mikkelsen B, Jensen J, Nielsen N (2004). “Elemental fingerprint analysis of barley (Hordeum vulgare) using inductively coupled plasma mass spectrometry, isotope-ratio mass spectrometry, and multivariate statistics”. 《Anal Bioanal Chem》 378 (1): 171–82. doi:10.1007/s00216-003-2219-0. PMID 14551660.
- ↑ Finney L, O'Halloran T (2003). “Transition metal speciation in the cell: insights from the chemistry of metal ion receptors”. 《Science》 300 (5621): 931–6. Bibcode:2003Sci...300..931F. doi:10.1126/science.1085049. PMID 12738850.
- ↑ Cousins R, Liuzzi J, Lichten L (2006). “Mammalian zinc transport, trafficking, and signals”. 《J Biol Chem》 281 (34): 24085–9. doi:10.1074/jbc.R600011200. PMID 16793761. 2008년 11월 5일에 원본 문서에서 보존된 문서. 2014년 10월 7일에 확인함.
- ↑ Dunn L, Rahmanto Y, Richardson D (2007). “Iron uptake and metabolism in the new millennium”. 《Trends Cell Biol》 17 (2): 93–100. doi:10.1016/j.tcb.2006.12.003. PMID 17194590.
- ↑ Nealson K, Conrad P (1999). “Life: past, present and future”. 《Philos Trans R Soc Lond B Biol Sci》 354 (1392): 1923–39. doi:10.1098/rstb.1999.0532. PMC 1692713. PMID 10670014.
- ↑ Nelson N, Ben-Shem A (2004). “The complex architecture of oxygenic photosynthesis”. 《Nat Rev Mol Cell Biol》 5 (12): 971–82. doi:10.1038/nrm1525. PMID 15573135.
- ↑ Häse C, Finkelstein R (December 1993). “Bacterial extracellular zinc-containing metalloproteases”. 《Microbiol Rev》 57 (4): 823–37. PMC 372940. PMID 8302217.
- ↑ Gupta R, Gupta N, Rathi P (2004). “Bacterial lipases: an overview of production, purification and biochemical properties”. 《Appl Microbiol Biotechnol》 64 (6): 763–81. doi:10.1007/s00253-004-1568-8. PMID 14966663.
- ↑ Hoyle T (1997). “The digestive system: linking theory and practice”. 《Br J Nurs》 6 (22): 1285–91. PMID 9470654.
- ↑ Souba W, Pacitti A (1992). “How amino acids get into cells: mechanisms, models, menus, and mediators”. 《JPEN J Parenter Enteral Nutr》 16 (6): 569–78. doi:10.1177/0148607192016006569. PMID 1494216.
- ↑ Barrett M, Walmsley A, Gould G (1999). “Structure and function of facilitative sugar transporters”. 《Curr Opin Cell Biol》 11 (4): 496–502. doi:10.1016/S0955-0674(99)80072-6. PMID 10449337.
- ↑ Bell G, Burant C, Takeda J, Gould G (1993). “Structure and function of mammalian facilitative sugar transporters”. 《J Biol Chem》 268 (26): 19161–4. PMID 8366068.
- ↑ 가 나 Bouché C, Serdy S, Kahn C, Goldfine A (2004). “The cellular fate of glucose and its relevance in type 2 diabetes”. 《Endocr Rev》 25 (5): 807–30. doi:10.1210/er.2003-0026. PMID 15466941. 2012년 12월 4일에 원본 문서에서 보존된 문서. 2014년 10월 7일에 확인함.
- ↑ Wipperman, Matthew, F.; Thomas, Suzanne, T.; Sampson, Nicole, S. (2014). “Pathogen roid rage: Cholesterol utilization by Mycobacterium tuberculosis”. 《Crit. Rev. Biochem. Mol. Biol.》. doi:10.3109/10409238.2014.895700. PMID 24611808.
- ↑ Sakami W, Harrington H (1963). “Amino acid metabolism”. 《Annu Rev Biochem》 32: 355–98. doi:10.1146/annurev.bi.32.070163.002035. PMID 14144484.
- ↑ Brosnan J (2000). “Glutamate, at the interface between amino acid and carbohydrate metabolism”. 《J Nutr》 130 (4S Suppl): 988S–90S. PMID 10736367.
- ↑ Young V, Ajami A (2001). “Glutamine: the emperor or his clothes?”. 《J Nutr》 131 (9 Suppl): 2449S–59S; discussion 2486S–7S. PMID 11533293.
- ↑ Hosler J, Ferguson-Miller S, Mills D (2006). “Energy Transduction: Proton Transfer Through the Respiratory Complexes”. 《Annu Rev Biochem》 75: 165–87. doi:10.1146/annurev.biochem.75.062003.101730. PMC 2659341. PMID 16756489.
- ↑ Schultz B, Chan S (2001). “Structures and proton-pumping strategies of mitochondrial respiratory enzymes”. 《Annu Rev Biophys Biomol Struct》 30: 23–65. doi:10.1146/annurev.biophys.30.1.23. PMID 11340051.
- ↑ Capaldi R, Aggeler R (2002). “Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor”. 《Trends Biochem Sci》 27 (3): 154–60. doi:10.1016/S0968-0004(01)02051-5. PMID 11893513.
- ↑ Friedrich B, Schwartz E (1993). “Molecular biology of hydrogen utilization in aerobic chemolithotrophs”. 《Annu Rev Microbiol》 47: 351–83. doi:10.1146/annurev.mi.47.100193.002031. PMID 8257102.
- ↑ Weber K, Achenbach L, Coates J (2006). “Microorganisms pumping iron: anaerobic microbial iron oxidation and reduction”. 《Nat Rev Microbiol》 4 (10): 752–64. doi:10.1038/nrmicro1490. PMID 16980937.
- ↑ Jetten M, Strous M, van de Pas-Schoonen K, Schalk J, van Dongen U, van de Graaf A, Logemann S, Muyzer G, van Loosdrecht M, Kuenen J (1998). “The anaerobic oxidation of ammonium”. 《FEMS Microbiol Rev》 22 (5): 421–37. doi:10.1111/j.1574-6976.1998.tb00379.x. PMID 9990725.
- ↑ Simon J (2002). “Enzymology and bioenergetics of respiratory nitrite ammonification”. 《FEMS Microbiol Rev》 26 (3): 285–309. doi:10.1111/j.1574-6976.2002.tb00616.x. PMID 12165429.
- ↑ Conrad R (1996). “Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O, and NO)”. 《Microbiol Rev》 60 (4): 609–40. PMC 239458. PMID 8987358.
- ↑ Barea J, Pozo M, Azcón R, Azcón-Aguilar C (2005). “Microbial co-operation in the rhizosphere”. 《J Exp Bot》 56 (417): 1761–78. doi:10.1093/jxb/eri197. PMID 15911555.
- ↑ van der Meer M, Schouten S, Bateson M, Nübel U, Wieland A, Kühl M, de Leeuw J, Sinninghe Damsté J, Ward D (July 2005). “Diel Variations in Carbon Metabolism by Green Nonsulfur-Like Bacteria in Alkaline Siliceous Hot Spring Microbial Mats from Yellowstone National Park”. 《Appl Environ Microbiol》 71 (7): 3978–86. doi:10.1128/AEM.71.7.3978-3986.2005. PMC 1168979. PMID 16000812.[깨진 링크(과거 내용 찾기)]
- ↑ Tichi M, Tabita F (2001). “Interactive Control of Rhodobacter capsulatus Redox-Balancing Systems during Phototrophic Metabolism”. 《J Bacteriol》 183 (21): 6344–54. doi:10.1128/JB.183.21.6344-6354.2001. PMC 100130. PMID 11591679.
- ↑ Allen J, Williams J (1998). “Photosynthetic reaction centers”. 《FEBS Lett》 438 (1–2): 5–9. doi:10.1016/S0014-5793(98)01245-9. PMID 9821949.
- ↑ Munekage Y, Hashimoto M, Miyake C, Tomizawa K, Endo T, Tasaka M, Shikanai T (2004). “Cyclic electron flow around photosystem I is essential for photosynthesis”. 《Nature》 429 (6991): 579–82. Bibcode:2004Natur.429..579M. doi:10.1038/nature02598. PMID 15175756.
- ↑ Miziorko H, Lorimer G (1983). “Ribulose-1,5-bisphosphate carboxylase-oxygenase”. 《Annu Rev Biochem》 52: 507–35. doi:10.1146/annurev.bi.52.070183.002451. PMID 6351728.
- ↑ Dodd A, Borland A, Haslam R, Griffiths H, Maxwell K (2002). “Crassulacean acid metabolism: plastic, fantastic”. 《J Exp Bot》 53 (369): 569–80. doi:10.1093/jexbot/53.369.569. PMID 11886877.
- ↑ Hügler M, Wirsen C, Fuchs G, Taylor C, Sievert S (May 2005). “Evidence for Autotrophic CO2 Fixation via the Reductive Tricarboxylic Acid Cycle by Members of the ɛ Subdivision of Proteobacteria”. 《J Bacteriol》 187 (9): 3020–7. doi:10.1128/JB.187.9.3020-3027.2005. PMC 1082812. PMID 15838028.
- ↑ Strauss G, Fuchs G (1993). “Enzymes of a novel autotrophic CO2 fixation pathway in the phototrophic bacterium Chloroflexus aurantiacus, the 3-hydroxypropionate cycle”. 《Eur J Biochem》 215 (3): 633–43. doi:10.1111/j.1432-1033.1993.tb18074.x. PMID 8354269.
- ↑ Wood H (1991). “Life with CO or CO2 and H2 as a source of carbon and energy”. 《FASEB J》 5 (2): 156–63. PMID 1900793.
- ↑ Shively J, van Keulen G, Meijer W (1998). “Something from almost nothing: carbon dioxide fixation in chemoautotrophs”. 《Annu Rev Microbiol》 52: 191–230. doi:10.1146/annurev.micro.52.1.191. PMID 9891798.
- ↑ Boiteux A, Hess B (1981). “Design of glycolysis”. 《Philos Trans R Soc Lond B Biol Sci》 293 (1063): 5–22. Bibcode:1981RSPTB.293....5B. doi:10.1098/rstb.1981.0056. PMID 6115423.
- ↑ Pilkis S, el-Maghrabi M, Claus T (1990). “Fructose-2,6-bisphosphate in control of hepatic gluconeogenesis. From metabolites to molecular genetics”. 《Diabetes Care》 13 (6): 582–99. doi:10.2337/diacare.13.6.582. PMID 2162755.
- ↑ 가 나 Ensign S (2006). “Revisiting the glyoxylate cycle: alternate pathways for microbial acetate assimilation”. 《Mol Microbiol》 61 (2): 274–6. doi:10.1111/j.1365-2958.2006.05247.x. PMID 16856935.
- ↑ Finn P, Dice J (2006). “Proteolytic and lipolytic responses to starvation”. 《Nutrition》 22 (7–8): 830–44. doi:10.1016/j.nut.2006.04.008. PMID 16815497.
- ↑ 가 나 Kornberg H, Krebs H (1957). “Synthesis of cell constituents from C2-units by a modified tricarboxylic acid cycle”. 《Nature》 179 (4568): 988–91. Bibcode:1957Natur.179..988K. doi:10.1038/179988a0. PMID 13430766.
- ↑ Rademacher T, Parekh R, Dwek R (1988). “Glycobiology”. 《Annu Rev Biochem》 57: 785–838. doi:10.1146/annurev.bi.57.070188.004033. PMID 3052290.
- ↑ Opdenakker G, Rudd P, Ponting C, Dwek R (1993). “Concepts and principles of glycobiology”. 《FASEB J》 7 (14): 1330–7. PMID 8224606.
- ↑ McConville M, Menon A (2000). “Recent developments in the cell biology and biochemistry of glycosylphosphatidylinositol lipids (review)”. 《Mol Membr Biol》 17 (1): 1–16. doi:10.1080/096876800294443. PMID 10824734.
- ↑ Chirala S, Wakil S (2004). “Structure and function of animal fatty acid synthase”. 《Lipids》 39 (11): 1045–53. doi:10.1007/s11745-004-1329-9. PMID 15726818.
- ↑ White S, Zheng J, Zhang Y (2005). “The structural biology of type II fatty acid biosynthesis”. 《Annu Rev Biochem》 74: 791–831. doi:10.1146/annurev.biochem.74.082803.133524. PMID 15952903.
- ↑ Ohlrogge J, Jaworski J (1997). “Regulation of fatty acid synthesis”. 《Annu Rev Plant Physiol Plant Mol Biol》 48: 109–136. doi:10.1146/annurev.arplant.48.1.109. PMID 15012259.
- ↑ Dubey V, Bhalla R, Luthra R (2003). “An overview of the non-mevalonate pathway for terpenoid biosynthesis in plants” (PDF). 《J Biosci》 28 (5): 637–46. doi:10.1007/BF02703339. PMID 14517367.
- ↑ 가 나 Kuzuyama T, Seto H (2003). “Diversity of the biosynthesis of the isoprene units”. 《Nat Prod Rep》 20 (2): 171–83. doi:10.1039/b109860h. PMID 12735695.
- ↑ Grochowski L, Xu H, White R (May 2006). “Methanocaldococcus jannaschii Uses a Modified Mevalonate Pathway for Biosynthesis of Isopentenyl Diphosphate”. 《J Bacteriol》 188 (9): 3192–8. doi:10.1128/JB.188.9.3192-3198.2006. PMC 1447442. PMID 16621811.
- ↑ Lichtenthaler H (1999). “The 1-Ddeoxy-D-xylulose-5-phosphate pathway of isoprenoid biosynthesis in plants”. 《Annu Rev Plant Physiol Plant Mol Biol》 50: 47–65. doi:10.1146/annurev.arplant.50.1.47. PMID 15012203.
- ↑ 가 나 Schroepfer G (1981). “Sterol biosynthesis”. 《Annu Rev Biochem》 50: 585–621. doi:10.1146/annurev.bi.50.070181.003101. PMID 7023367.
- ↑ Lees N, Skaggs B, Kirsch D, Bard M (1995). “Cloning of the late genes in the ergosterol biosynthetic pathway of Saccharomyces cerevisiae—a review”. 《Lipids》 30 (3): 221–6. doi:10.1007/BF02537824. PMID 7791529.
- ↑ Himmelreich R, Hilbert H, Plagens H, Pirkl E, Li BC, Herrmann R (November 1996). “Complete sequence analysis of the genome of the bacterium Mycoplasma pneumoniae”. 《Nucleic Acids Res.》 24 (22): 4420–49. doi:10.1093/nar/24.22.4420. PMC 146264. PMID 8948633.
- ↑ Guyton, Arthur C.; John E. Hall (2006). 《Textbook of Medical Physiology》. Philadelphia: Elsevier. 855–6쪽. ISBN 0-7216-0240-1.
- ↑ Ibba M, Söll D (2001). “The renaissance of aminoacyl-tRNA synthesis”. 《EMBO Rep》 2 (5): 382–7. doi:10.1093/embo-reports/kve095. PMC 1083889. PMID 11375928. 2011년 5월 1일에 원본 문서에서 보존된 문서. 2017년 5월 20일에 확인함.
- ↑ Lengyel P, Söll D (1969). “Mechanism of protein biosynthesis”. 《Bacteriol Rev》 33 (2): 264–301. PMC 378322. PMID 4896351.
- ↑ 가 나 Rudolph F (1994). “The biochemistry and physiology of nucleotides”. 《J Nutr》 124 (1 Suppl): 124S–127S. PMID 8283301. Zrenner R, Stitt M, Sonnewald U, Boldt R (2006). “Pyrimidine and purine biosynthesis and degradation in plants”. 《Annu Rev Plant Biol》 57: 805–36. doi:10.1146/annurev.arplant.57.032905.105421. PMID 16669783.
- ↑ Stasolla C, Katahira R, Thorpe T, Ashihara H (2003). “Purine and pyrimidine nucleotide metabolism in higher plants”. 《J Plant Physiol》 160 (11): 1271–95. doi:10.1078/0176-1617-01169. PMID 14658380.
- ↑ Davies O, Mendes P, Smallbone K, Malys N (2012). “Characterisation of multiple substrate-specific (d)ITP/(d)XTPase and modelling of deaminated purine nucleotide metabolism”. 《BMB Reports》 45 (4): 259–64. doi:10.5483/BMBRep.2012.45.4.259. PMID 22531138.
- ↑ Smith J (1995). “Enzymes of nucleotide synthesis”. 《Curr Opin Struct Biol》 5 (6): 752–7. doi:10.1016/0959-440X(95)80007-7. PMID 8749362.
- ↑ Testa B, Krämer S (2006). “The biochemistry of drug metabolism—an introduction: part 1. Principles and overview”. 《Chem Biodivers》 3 (10): 1053–101. doi:10.1002/cbdv.200690111. PMID 17193224.
- ↑ Danielson P (2002). “The cytochrome P450 superfamily: biochemistry, evolution and drug metabolism in humans”. 《Curr Drug Metab》 3 (6): 561–97. doi:10.2174/1389200023337054. PMID 12369887.
- ↑ King C, Rios G, Green M, Tephly T (2000). “UDP-glucuronosyltransferases”. 《Curr Drug Metab》 1 (2): 143–61. doi:10.2174/1389200003339171. PMID 11465080.
- ↑ Sheehan D, Meade G, Foley V, Dowd C (November 2001). “Structure, function and evolution of glutathione transferases: implications for classification of non-mammalian members of an ancient enzyme superfamily”. 《Biochem J》 360 (Pt 1): 1–16. doi:10.1042/0264-6021:3600001. PMC 1222196. PMID 11695986.
- ↑ Galvão T, Mohn W, de Lorenzo V (2005). “Exploring the microbial biodegradation and biotransformation gene pool”. 《Trends Biotechnol》 23 (10): 497–506. doi:10.1016/j.tibtech.2005.08.002. PMID 16125262.
- ↑ Janssen D, Dinkla I, Poelarends G, Terpstra P (2005). “Bacterial degradation of xenobiotic compounds: evolution and distribution of novel enzyme activities”. 《Environ Microbiol》 7 (12): 1868–82. doi:10.1111/j.1462-2920.2005.00966.x. PMID 16309386.
- ↑ Davies K (1995). “Oxidative stress: the paradox of aerobic life”. 《Biochem Soc Symp》 61: 1–31. PMID 8660387.
- ↑ Tu B, Weissman J (2004). “Oxidative protein folding in eukaryotes: mechanisms and consequences”. 《J Cell Biol》 164 (3): 341–6. doi:10.1083/jcb.200311055. PMC 2172237. PMID 14757749.
- ↑ Sies H (1997). “Oxidative stress: oxidants and antioxidants” (PDF). 《Exp Physiol》 82 (2): 291–5. PMID 9129943. 2009년 3월 25일에 원본 문서 (PDF)에서 보존된 문서. 2014년 10월 7일에 확인함.
- ↑ Vertuani S, Angusti A, Manfredini S (2004). “The antioxidants and pro-antioxidants network: an overview”. 《Curr Pharm Des》 10 (14): 1677–94. doi:10.2174/1381612043384655. PMID 15134565.
- ↑ von Stockar U, Liu J (1999). “Does microbial life always feed on negative entropy? Thermodynamic analysis of microbial growth”. 《Biochim Biophys Acta》 1412 (3): 191–211. doi:10.1016/S0005-2728(99)00065-1. PMID 10482783.
- ↑ Demirel Y, Sandler S (2002). “Thermodynamics and bioenergetics”. 《Biophys Chem》 97 (2–3): 87–111. doi:10.1016/S0301-4622(02)00069-8. PMID 12050002.
- ↑ Albert R (2005). “Scale-free networks in cell biology”. 《J Cell Sci》 118 (Pt 21): 4947–57. doi:10.1242/jcs.02714. PMID 16254242.
- ↑ Brand M (1997). “Regulation analysis of energy metabolism”. 《J Exp Biol》 200 (Pt 2): 193–202. PMID 9050227.
- ↑ Soyer O, Salathé M, Bonhoeffer S (2006). “Signal transduction networks: topology, response and biochemical processes”. 《J Theor Biol》 238 (2): 416–25. doi:10.1016/j.jtbi.2005.05.030. PMID 16045939.
- ↑ 가 나 Salter M, Knowles R, Pogson C (1994). “Metabolic control”. 《Essays Biochem》 28: 1–12. PMID 7925313.
- ↑ Westerhoff H, Groen A, Wanders R (1984). “Modern theories of metabolic control and their applications (review)”. 《Biosci Rep》 4 (1): 1–22. doi:10.1007/BF01120819. PMID 6365197.
- ↑ Fell D, Thomas S (1995). “Physiological control of metabolic flux: the requirement for multisite modulation”. 《Biochem J》 311 (Pt 1): 35–9. PMC 1136115. PMID 7575476.
- ↑ Hendrickson W (2005). “Transduction of biochemical signals across cell membranes”. 《Q Rev Biophys》 38 (4): 321–30. doi:10.1017/S0033583506004136. PMID 16600054.
- ↑ Cohen P (2000). “The regulation of protein function by multisite phosphorylation—a 25 year update”. 《Trends Biochem Sci》 25 (12): 596–601. doi:10.1016/S0968-0004(00)01712-6. PMID 11116185.
- ↑ Lienhard G, Slot J, James D, Mueckler M (1992). “How cells absorb glucose”. 《Sci Am》 266 (1): 86–91. doi:10.1038/scientificamerican0192-86. PMID 1734513.
- ↑ Roach P (2002). “Glycogen and its metabolism”. 《Curr Mol Med》 2 (2): 101–20. doi:10.2174/1566524024605761. PMID 11949930.
- ↑ Newgard C, Brady M, O'Doherty R, Saltiel A (2000). “Organizing glucose disposal: emerging roles of the glycogen targeting subunits of protein phosphatase-1” (PDF). 《Diabetes》 49 (12): 1967–77. doi:10.2337/diabetes.49.12.1967. PMID 11117996.
- ↑ Romano A, Conway T (1996). “Evolution of carbohydrate metabolic pathways”. 《Res Microbiol》 147 (6–7): 448–55. doi:10.1016/0923-2508(96)83998-2. PMID 9084754.
- ↑ Koch A (1998). “How did bacteria come to be?”. 《Adv Microb Physiol》. Advances in Microbial Physiology 40: 353–99. doi:10.1016/S0065-2911(08)60135-6. ISBN 978-0-12-027740-7. PMID 9889982.
- ↑ Ouzounis C, Kyrpides N (1996). “The emergence of major cellular processes in evolution”. 《FEBS Lett》 390 (2): 119–23. doi:10.1016/0014-5793(96)00631-X. PMID 8706840.
- ↑ C.Michael Hogan. 2010. Mutation. ed. E.Monosson and C.J.Cleveland. Encyclopedia of Earth. National Council for Science and the Environment. Washington DC
- ↑ Caetano-Anolles G, Kim HS, Mittenthal JE (2007). “The origin of modern metabolic networks inferred from phylogenomic analysis of protein architecture”. 《Proc Natl Acad Sci USA》 104 (22): 9358–63. Bibcode:2007PNAS..104.9358C. doi:10.1073/pnas.0701214104. PMC 1890499. PMID 17517598.
- ↑ Schmidt S, Sunyaev S, Bork P, Dandekar T (2003). “Metabolites: a helping hand for pathway evolution?”. 《Trends Biochem Sci》 28 (6): 336–41. doi:10.1016/S0968-0004(03)00114-2. PMID 12826406.
- ↑ Light S, Kraulis P (2004). “Network analysis of metabolic enzyme evolution in Escherichia coli”. 《BMC Bioinformatics》 5: 15. doi:10.1186/1471-2105-5-15. PMC 394313. PMID 15113413.
- ↑ Alves R, Chaleil R, Sternberg M (2002). “Evolution of enzymes in metabolism: a network perspective”. 《J Mol Biol》 320 (4): 751–70. doi:10.1016/S0022-2836(02)00546-6. PMID 12095253.
- ↑ Kim HS, Mittenthal JE, Caetano-Anolles G (2006). “MANET: tracing evolution of protein architecture in metabolic networks”. 《BMC Bioinformatics》 7: 351. doi:10.1186/1471-2105-7-351. PMC 1559654. PMID 16854231.
- ↑ Teichmann SA, Rison SC, Thornton JM, Riley M, Gough J, Chothia C (2001). “Small-molecule metabolsim: an enzyme mosaic”. 《Trends Biotechnol》 19 (12): 482–6. doi:10.1016/S0167-7799(01)01813-3. PMID 11711174.
- ↑ Spirin V, Gelfand M, Mironov A, Mirny L (June 2006). “A metabolic network in the evolutionary context: Multiscale structure and modularity”. 《Proc Natl Acad Sci USA》 103 (23): 8774–9. Bibcode:2006PNAS..103.8774S. doi:10.1073/pnas.0510258103. PMC 1482654. PMID 16731630.
- ↑ Lawrence J (2005). “Common themes in the genome strategies of pathogens”. 《Curr Opin Genet Dev》 15 (6): 584–8. doi:10.1016/j.gde.2005.09.007. PMID 16188434.
- ↑ Wernegreen J (2005). “For better or worse: genomic consequences of intracellular mutualism and parasitism”. 《Curr Opin Genet Dev》 15 (6): 572–83. doi:10.1016/j.gde.2005.09.013. PMID 16230003.
- ↑ Pál C, Papp B, Lercher M, Csermely P, Oliver S, Hurst L (2006). “Chance and necessity in the evolution of minimal metabolic networks”. 《Nature》 440 (7084): 667–70. Bibcode:2006Natur.440..667P. doi:10.1038/nature04568. PMID 16572170.
- ↑ Rennie M (1999). “An introduction to the use of tracers in nutrition and metabolism”. 《Proc Nutr Soc》 58 (4): 935–44. doi:10.1017/S002966519900124X. PMID 10817161.
- ↑ Phair R (1997). “Development of kinetic models in the nonlinear world of molecular cell biology”. 《Metabolism》 46 (12): 1489–95. doi:10.1016/S0026-0495(97)90154-2. PMID 9439549.
- ↑ Sterck L, Rombauts S, Vandepoele K, Rouzé P, Van de Peer Y (2007). “How many genes are there in plants (... and why are they there)?”. 《Curr Opin Plant Biol》 10 (2): 199–203. doi:10.1016/j.pbi.2007.01.004. PMID 17289424.
- ↑ Borodina I, Nielsen J (2005). “From genomes to in silico cells via metabolic networks”. 《Curr Opin Biotechnol》 16 (3): 350–5. doi:10.1016/j.copbio.2005.04.008. PMID 15961036.
- ↑ Gianchandani E, Brautigan D, Papin J (2006). “Systems analyses characterize integrated functions of biochemical networks”. 《Trends Biochem Sci》 31 (5): 284–91. doi:10.1016/j.tibs.2006.03.007. PMID 16616498.
- ↑ Duarte NC, Becker SA, Jamshidi N; 외. (February 2007). “Global reconstruction of the human metabolic network based on genomic and bibliomic data”. 《Proc. Natl. Acad. Sci. U.S.A.》 104 (6): 1777–82. Bibcode:2007PNAS..104.1777D. doi:10.1073/pnas.0610772104. PMC 1794290. PMID 17267599.
- ↑ Goh KI, Cusick ME, Valle D, Childs B, Vidal M, Barabási AL (May 2007). “The human disease network”. 《Proc. Natl. Acad. Sci. U.S.A.》 104 (21): 8685–90. Bibcode:2007PNAS..104.8685G. doi:10.1073/pnas.0701361104. PMC 1885563. PMID 17502601.
- ↑ Lee DS, Park J, Kay KA, Christakis NA, Oltvai ZN, Barabási AL (July 2008). “The implications of human metabolic network topology for disease comorbidity”. 《Proc. Natl. Acad. Sci. U.S.A.》 105 (29): 9880–9885. Bibcode:2008PNAS..105.9880L. doi:10.1073/pnas.0802208105. PMC 2481357. PMID 18599447.
- ↑ Csete M, Doyle J (2004). “Bow ties, metabolism and disease”. 《Trends Biotechnol.》 22 (9): 446–50. doi:10.1016/j.tibtech.2004.07.007. PMID 15331224.
- ↑ Ma HW, Zeng AP (2003). “The connectivity structure, giant strong component and centrality of metabolic networks”. 《Bioinformatics》 19 (11): 1423–30. doi:10.1093/bioinformatics/btg177. PMID 12874056.
- ↑ Zhao J, Yu H, Luo JH, Cao ZW, Li YX (2006). “Hierarchical modularity of nested bow-ties in metabolic networks”. 《BMC Bioinformatics》 7: 386. doi:10.1186/1471-2105-7-386. PMC 1560398. PMID 16916470.
- ↑ Thykaer J, Nielsen J (2003). “Metabolic engineering of beta-lactam production”. 《Metab Eng》 5 (1): 56–69. doi:10.1016/S1096-7176(03)00003-X. PMID 12749845.
- ↑ González-Pajuelo M, Meynial-Salles I, Mendes F, Andrade J, Vasconcelos I, Soucaille P (2005). “Metabolic engineering of Clostridium acetobutylicum for the industrial production of 1,3-propanediol from glycerol”. 《Metab Eng》 7 (5–6): 329–36. doi:10.1016/j.ymben.2005.06.001. PMID 16095939.
- ↑ Krämer M, Bongaerts J, Bovenberg R, Kremer S, Müller U, Orf S, Wubbolts M, Raeven L (2003). “Metabolic engineering for microbial production of shikimic acid”. 《Metab Eng》 5 (4): 277–83. doi:10.1016/j.ymben.2003.09.001. PMID 14642355.
- ↑ Koffas M, Roberge C, Lee K, Stephanopoulos G (1999). “Metabolic engineering”. 《Annu Rev Biomed Eng》 1: 535–57. doi:10.1146/annurev.bioeng.1.1.535. PMID 11701499.
- ↑ “Metabolism”. The Online Etymology Dictionary. 2007년 2월 20일에 확인함.
- ↑ Dr. Abu Shadi Al-Roubi (1982), "Ibn Al-Nafis as a philosopher", Symposium on Ibn al-Nafis, Second International Conference on Islamic Medicine: Islamic Medical Organization, Kuwait (cf. Ibn al-Nafis As a Philosopher, Encyclopedia of Islamic World [1])
- ↑ Eknoyan G (1999). “Santorio Sanctorius (1561–1636) – founding father of metabolic balance studies”. 《Am J Nephrol》 19 (2): 226–33. doi:10.1159/000013455. PMID 10213823.
- ↑ Williams, H. S. (1904) A History of Science: in Five Volumes. Volume IV: Modern Development of the Chemical and Biological Sciences Harper and Brothers (New York) Retrieved on 2007-03-26
- ↑ Dubos J. (1951). “Louis Pasteur: Free Lance of Science, Gollancz. Quoted in Manchester K. L. (1995) Louis Pasteur (1822–1895)—chance and the prepared mind”. 《Trends Biotechnol》 13 (12): 511–515. doi:10.1016/S0167-7799(00)89014-9. PMID 8595136.
- ↑ Kinne-Saffran E, Kinne R (1999). “Vitalism and synthesis of urea. From Friedrich Wöhler to Hans A. Krebs”. 《Am J Nephrol》 19 (2): 290–4. doi:10.1159/000013463. PMID 10213830.
- ↑ Eduard Buchner's 1907 Nobel lecture at http://nobelprize.org Accessed 2007-03-20
- ↑ Kornberg H (2000). “Krebs and his trinity of cycles”. 《Nat Rev Mol Cell Biol》 1 (3): 225–8. doi:10.1038/35043073. PMID 11252898.
- ↑ Krebs HA, Henseleit K (1932). “Untersuchungen über die Harnstoffbildung im tierkorper”. 《Z. Physiol. Chem.》 210: 33–66. doi:10.1515/bchm2.1932.210.1-2.33.
- ↑ Krebs H, Johnson W (April 1937). “Metabolism of ketonic acids in animal tissues”. 《Biochem J》 31 (4): 645–60. PMC 1266984. PMID 16746382.
