퓨린 대사
퓨린 대사(영어: purine metabolism)는 많은 생물체에 존재하는 퓨린을 합성하고 분해하는 대사 경로를 의미한다.
생합성
[편집]퓨린은 생물학적으로 뉴클레오타이드로 합성되며, 특히 리보뉴클레오타이드(즉, 핵염기, 리보스, 인산으로 구성됨)로 합성된다. 아데노신 일인산(AMP)과 구아노신 일인산(GMP)은 둘 다 이노신 일인산(IMP)으로부터 유도된 것이며, 이노신 일인산은 퓨린 뉴클레오타이드 생합성 경로에서 완전한 퓨린 고리를 가지고 있는 첫 번째 화합물이다.
IMP의 합성
[편집]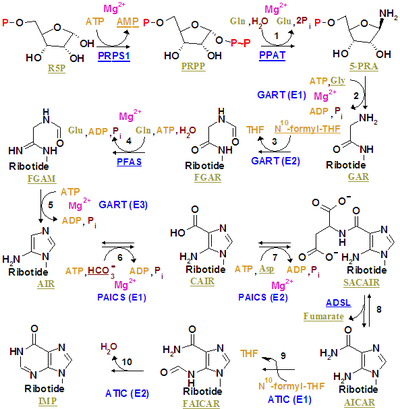
이노신 일인산(IMP)은 복잡한 대사 경로(오른쪽 그림을 참조)를 통해 리보스 5-인산으로부터 합성된다. 퓨린 고리를 구성하는 5개의 탄소 원자와 4개의 질소 원자의 기원은 아스파르트산, CO2, 글리신, 폼산, 글루타민과 같은 여러 분자들로부터 유래한다. 보다 구체적으로 퓨린 고리를 구성하는 원자들은 글리신으로부터 2개의 탄소 원자와 1개의 질소 원자, 글루타민으로부터 2개의 질소 원자, 아스파르트산으로부터 1개의 질소 원자, 폼산(10-폼일테트라하이드로폴산의 형태로 공급됨)으로부터 2개의 탄소 원자, 탄산수소염으로부터 1개의 탄소 원자가 유래하였다. 폼일기로부터 유래하는 2개의 탄소 원자는 퓨린 고리 시스템에서 두 질소 원자 사이에 다리 역할을 하는 2번 탄소와 8번 탄소를 구성한다.
핵심적인 조절 단계는 리보스-인산 다이포스포키네이스에 의한 리보스 5-인산으로부터 포스포리보실 피로인산(PRPP)의 생성 단계이며, 리보스-인산 다이포스포키네이스는 무기 인산에 의해 활성화되고 퓨린 뉴클레오타이드에 의해 불활성화된다. 포스포리보실 피로인산은 퓨린 합성 뿐만 아니라 피리미딘 합성 및 회수 경로에서도 사용된다.
첫 번째 개입 단계(committed step)는 포스포리보실 피로인산(PRPP), 글루타민이 아미도포스포리보실트랜스퍼레이스(글루타민-PRPP 아미도트랜스퍼레이스)에 의해 포스포리보실아민, 글루탐산, 피로인산으로 전환되는 것이다. 아미도포스포리보실트랜스퍼레이스는 포스포리보실 피로인산에 의해 활성화되고, AMP, GMP, IMP에 의해 저해된다.
- 포스포리보실 피로인산(PRPP) + L-글루타민 + H2O → 포스포리보실아민(PRA) + L-글루탐산 + PPi
두 번째 단계에서 포스포리보실아민, 글리신, ATP는 포스포리보실아민-글리신 연결효소(GAR 합성효소)에 의해 글리신아마이드 리보뉴클레오타이드(GAR), ADP, Pi로 전환된다. pH 7.5, 37 °C에서 반감기가 38초인 포스포리보실아민(PRA)의 화학적 불안정성 때문에 연구자들은 포스포리보실아민이 생체 내에서 아미도포스포리보실트랜스퍼레이스로부터 GAR 합성효소로 보내진다고 제안했다.[1]
- 포스포리보실아민(PRA) + 글리신 + ATP → 글리신아마이드 리보튜클레오타이드(GAR) + ADP + Pi
세 번째 단계에서 글리신아마이드 리보뉴클레오타이드(GAR), 10-폼일테트라하이드로폴산은 포스포리보실글리신아마이드 폼일트랜스퍼레이스(GAR 트랜스폼일레이스)에 의해 폼일글리신아마이드 리보뉴클레오타이드(FGAR), 테트라하이드로폴산으로 전환된다.
글리신아마이드 리보뉴클레오타이드(GAR) + 10-폼일테트라하이드로폴산(fTHF) → 폼일글리신아마이드 리보뉴클레오타이드(fGAR) + 테트라하이드로폴산(THF)
네 번째 단계에서 폼일글리신아마이드 리보뉴클레오타이드(fGAR), 글루타민, ATP는 포스포리보실폼일글리신아미딘 생성효소(fGAR 아미도트랜스퍼레이스)에 의해 폼일글리신아미딘 리보뉴클레오타이드(fGAM), 글루탐산, ADP, Pi로 전환된다.
- 폼일글리신아마이드 리보뉴클레오타이드(fGAR) + L-글루타민 + ATP → 폼일글리신아미딘 리보뉴클레오타이드(fGAM) + L-글루탐산 + ADP + Pi
다섯 번째 단계에서 폼일글리신아미딘 리보뉴클레오타이드(fGAM), ATP는 AIR 합성효소(fGAM 고리화효소)에 의해 5-아미노이미다졸 리보뉴클레오타이드(AIR), ADP, Pi로 전환된다.
- 폼일글리신아미딘 리보뉴클레오타이드(fGAM) + ATP → 5-아미노이미다졸 리보뉴클레오타이드(AIR) + ADP + Pi + H2O
여섯 번째 단계에서 5-아미노이미다졸 리보뉴클레오타이드(AIR), CO2는 포스포리보실아미노이미다졸 카복실화효소(AIR 카복실화효소)에 의해 카복시아미노이미다졸 리보뉴클레오타이드(CAIR)로 전환된다.
- 5-아미노이미다졸 리보뉴클레오타이드(AIR) + CO2 → 카복시아미노이미다졸 리보뉴클레오타이드(CAIR) + 2H+
일곱 번째 단계에서 카복시아미노이미다졸 리보뉴클레오타이드(CAIR), 아스파르트산, ATP는 포스포리보실아미노이미다졸석시노카복사마이드 생성효소(SAICAR 합성효소)에 의해 N-석시닐-5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(SAICAR), ADP, Pi로 전환된다.
- 카복시아미노이미다졸(CAIR) + L-아스파르트산 + ATP → N-석시닐-5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(SAICAR) + ADP + Pi
여덟 번째 단계에서 N-석시닐-5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(SAICAR)는 아데닐로석신산 분해효소(SAICAR 분해효소)에 의해 5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(AICAR), 푸마르산으로 전환된다.
- N-석시닐-5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(SAICAR) → 5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(AICAR) + 푸마르산
5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(AICAR)와 푸마르산은 두 가지 다른 경로로 들어간다. 5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드는 아홉 번째 단계의 반응물로 사용되는 반면에 푸마르산은 시트르산 회로로 들어가서 말산으로 전환될 수 있다. 푸마르산의 말산으로의 전환은 푸마레이스에 의해 촉매된다. 이러한 방법으로 푸마르산은 퓨린 합성을 시트르산 회로와 연결시킨다.[2]
아홉 번째 단계에서 5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(AICAR), 10-폼일테트라하이드로폴산은 포스포리보실아미노이미다졸카복사마이드 폼일트랜스퍼레이스(AICAR 트랜스폼일레이스)에 의해 N-폼일아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(FAICAR), 테트라하이드로폴산으로 전환된다.
- 5-아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(AICAR) + 10-폼일테트라하이드로폴산(fTHF) → N-폼일아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(FAICAR) + 테트라하이드로폴산(THF)
마지막 단계에서 N-폼일아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(FAICAR)는 이노신 일인산 생성효소(IMP 생성효소)에 의해 이노신 일인산(IMP)와 H2O로 전환된다.
- N-폼일아미노이미다졸-4-카복사마이드 리보뉴클레오타이드(FAICAR) → 이노신 일인산(IMP) + H2O
진핵생물에서 두 번째, 세 번째 및 다섯 번째 단계는 GART 유전자에 의해 암호화되는 삼기능성 퓨린 생합성 단백질 아데노신-3에 의해 촉매된다.
아홉 번째 단계와 열 번째 단계는 둘 다 ATIC 유전자에 의해 암호화되는 이기능성 퓨린 생합성 단백질 PURH라는 단일 단백질에 의해 수행된다.
IMP로부터 GMP의 생합성
[편집]- 이노신 일인산(IMP) + NAD+ → 잔토신 일인산(XMP) + NADH + H+
- 잔토신 일인산(XMP) + 글루타민 + ATP → 구아노신 일인산(GMP) + 글루탐산 + AMP + PPi
IMP로부터 AMP의 생합성
[편집]- 이노신 일인산(IMP)은 아데닐로석신산 생성효소(아데닐로석신산 합성효소)에 의해 아데닐로석신산으로 전환된다.
- 이노신 일인산(IMP) + 아스파르트산 + GTP → 아데닐로석신산 + GDP + Pi
- 아데닐로석신산은 아데놀로석신산 분해효소에 의해 아데노신 일인산(AMP)과 푸마르산으로 전환된다.
분해
[편집]퓨린은 여러 효소들에 의해서 분해 대사된다.
GMP의 분해
[편집]- 구아노신 일인산(GMP)은 뉴클레오타이드 가수분해효소에 의해 구아노신으로 전환된다.
- 구아노신 일인산(GMP) + H2O → 구아노신 + Pi
- 구아노신은 뉴클레오사이드 가수분해효소에 의해 구아닌으로 전환된다.
- 구아노신 + H2O → 구아닌 + 리보스
- 구아닌은 구아닌 탈아미노효소에 의해 잔틴으로 전환된다.
- 구아닌 + H2O → 잔틴 + NH3
- 잔틴 + H2O + Osub>2 → 요산 + H2O2
AMP의 분해
[편집]- 아데노신 일인산(AMP)은 뉴클레오타이드 가수분해효소에 의해 아데노신으로 전환된다.
- 아데노신 일인산(AMP) + H2O → 아데노신 + Pi
- 아데노신은 아데노신 탈아미노효소에 의해 이노신으로 전환된다.
- 아데노신 + H2O → 이노신 + NH3
- 이노신은 뉴클레오사이드 가수분해효소에 의해 하이포잔틴으로 전환된다.
- 이노신 + H2O → 하이포잔틴 + 리보스
- 하이포잔틴 + H2O + O2 → 잔틴 + H2O2
- 잔틴은 잔틴 산화효소에 의해 요산으로 전환된다.
- 잔틴 + H2O + O2 → 요산 + H2O2
퓨린 뉴클레오타이드 생합성의 조절
[편집]포스포리보실 피로인산(PRPP)이 아미도포스포리보실트랜스퍼레이스(글루타민-PRPP 아미도트랜스퍼레이스)에 의해 포스포리보실아민으로 전환되는 단계는 퓨린 생합성의 조절 지점이다. 아미도포스포리보실트랜스퍼레이스는 다른 자리 입체성 조절 효소이고, IMP, GMP, AMP는 이 효소의 저해제이며, 포스포리보실 피로인산(PRPP)은 활성제이다.
회수 경로
[편집]세포 내 핵산의 전환으로 생성된(또는 음식으로 섭취된) 퓨린은 회수되어 새로운 뉴클레오타이드에서 재사용될 수 있다.
- 아데닌 포스포리보실트랜스퍼레이스(APRT)는 아데닌을 회수한다.
- 아데닌 + 포스포리보실 피로인산(PRPP) → 아데노신 일인산(AMP) + PPi
- 하이포잔틴-구아닌 포스포리보실트랜스퍼레이스(HGPRT)는 구아닌과 하이포잔틴을 회수한다.[3] 하이포잔틴-구아닌 포스포리보실트랜스퍼레이스의 유전적 결핍은 레쉬-니한 증후군을 일으킨다.
대사 이상
[편집]퓨린과 피리미딘의 대사에 관여하는 유전자에 이상이 생기면, 유전 질환으로 고통을 겪을 수 있다. 이러한 퓨린과 피리미딘 대사 이상 질환의 증상으로는 통풍, 빈혈, 간질, 발달 지연, 난청, 강박 관념, 신장 이상 또는 신장 결석, 면역력 상실 등이 있을 수 있다.
퓨린 대사의 이상은 DNA와 RNA에 삽입될 수 있는 유해한 뉴클레오사이드 삼인산으로부터 발생할 수 있는 유전 정보의 균형을 깨뜨릴 수 있으며, 이로 인해 유전적 장애와 변이를 더욱 초래하고, 그 결과 여러 종류의 질병들을 발생시킬 수 있다. 이러한 질병들 중 일부는 다음과 같다.
- 아데노신 탈아미노효소의 소실에 의한 중증 면역결핍증.
- 하이포잔틴-구아닌 포스포리보실트랜스퍼레이스의 소실에 의한 고요산혈증 및 레쉬-니한 증후군.
- IMP 탈수소효소와 같은 효소의 활성이 증가함에 따라 다른 종류의 암의 발생.[4]
약물 요법
[편집]퓨린 대사의 조절은 약물치료적 가치를 가지고 있다.
퓨린 합성 저해제는 세포, 특히 백혈구의 증식을 저해한다. 이러한 저해제로는 장기 이식, 류머티스 관절염과 같은 자가면역질환, 크론병 및 궤양성 대장염과 같은 염증성 장질환에 사용되는 면역억제제인 아자티오프린이 있다.
마이코페놀산은 장기 이식에서 거부 반응을 막기 위해 사용되는 면역 억제제이다. 마이코페놀산은 이노시톨 일인산 탈수소효소를 차단함으로써 퓨린 합성을 저해한다. 또한 메토트렉세이트는 폴산의 대사를 차단함으로써 간접적으로 퓨린 합성을 저해한다(메토트렉세이트는 다이하이드로폴산 환원효소의 저해제이다).
알로퓨리놀은 잔틴 산화효소를 저해해서 체내 요산의 수준을 낮추는 약물이다. 알로퓨리놀은 과도한 요산에 의해 관절에 결정이 형성되는 질병인 통풍의 치료에 유용하다.
같이 보기
[편집]- 피리미딘 대사
- 퓨린
- 퓨린성 신호전달
- 질병 완화 항류마티스제 (DMARD)
각주
[편집]- ↑ Antle, Vincent D.; Liu, Dashan; McKellar, B. Robert; Caperelli, Carol A.; Hua, Mei; Vince, Robert (1996년 4월 5일). “Substrate specificity of glycinamide ribonucleotide synthetase from chicken liver”. 《The Journal of Biological Chemistry》 271 (14): 8192–5. doi:10.1074/jbc.271.14.8192. PMID 8626510.
- ↑ H., Garrett, Reginald (2016년 2월 11일). 《Biochemistry》. Grisham, Charles M. Six판. Boston, MA. 666 & 934쪽. ISBN 9781305577206. OCLC 914290655.
- ↑ Ansari MY, Equbal A, Dikhit MR, Mansuri R, Rana S, Ali V, Sahoo GC, Das P (Nov 2015). “Establishment of Correlation between In-Silico &In-Vitro Test Analysis against Leishmania HGPRT to inhibitors”. 《International Journal of Biological Macromolecules》 83: 78–96. doi:10.1016/j.ijbiomac.2015.11.051. PMID 26616453.
- ↑ Pang, Bo; McFaline, Jose L.; Burgis, Nicholas E.; Dong, Min; Taghizadeh, Koli; Sullivan, Matthew R.; Elmquist, C. Eric; Cunningham, Richard P.; Dedon, Peter C. (2012). “Defects in purine nucleotide metabolism lead to substantial incorporation of xanthine and hypoxanthine into DNA and RNA”. 《Proceedings of the National Academy of Sciences of the United States of America》 109 (7): 2319–2324. Bibcode:2012PNAS..109.2319P. doi:10.1073/pnas.1118455109. JSTOR 41477470. PMC 3289290. PMID 22308425.
외부 링크
[편집]- The Medical Biochemistry Page
- Purine metabolism - Reference pathway Archived 2020년 9월 21일 - 웨이백 머신
- PUMPA: Purine Metabolic Patients’ Association Archived 2020년 11월 29일 - 웨이백 머신
