SEIR 모형
SEIR 모형은 역학에서 전염병의 확산을 설명할 때 수학적인 모델링을 통해 접근할 수 있는 방법 중 하나이다. 이 모형은 SIR 모형보다는 더 현실에 가깝다고 여겨지며, 이는 병에 막 감염된 개체가 당장은 다른 개체에게 병을 옮기지 않는다는 점을 고려하기 때문이다. 개체에 기반한 모형보다는 거시적으로 개체군 전체를 조명하는 방식에 가깝다.
방정식
[편집]N 개체로 이루어진 개체군을 S, E, I, R의 네 그룹으로 나누어
혹은
의 공식이 성립되도록 한다.
모든 개체는
- Susceptible (S, 감염대상군) → Exposed (E, 접촉군) → Infectious (I, 감염군) → Recovered (R, 회복군)
의 단계를 거친다. 전염병의 확산은 다음과 같이 설명될 수 있다.
이것은 상미분방정식의 비선형 시스템에 속한다. 문헌에서는 여러 형태의 공식을 발견할 수 있는데 예를 들면 항을 대신 쓰는 식이다. 이외에도 의 합을 반드시 이라고 정하는 대신 N을 전체 개체군이라 할 때 라고 정의할 수도 있다.
| 수량 | 단위 | 설명 |
|---|---|---|
| S(t) | 1 | 감염대상군 (Susceptible): 감염될 수 있으나 아직 감염되지 않은 개체의 수 |
| E(t) | 1 | 접촉군 (Exposed): 이미 감염되었으나 아직 병을 전염시키지는 않는 단계에 있는 개체의 수 |
| I(t) | 1 | 감염군 (Infectious): 병을 전염시킬 수 있는 개체의 수 |
| R(t) | 1 | 회복군 (Recovered 또는 Resistent): 병에서 회복한 개체의 수와 격리 중 사망한 개체의 수의 합 |
| t | d | 시간 (일) |
| β | 1/d | 감염률 (Transmission rate). 역수는 접촉 평균 시간과 같다. |
| γ | 1/d | 회복률 (Recovery rate). 역수는 평균 전염 가능 시간과 같다. |
| a | 1/d | 역수는 평균 지연 시간 (latency)과 같다. |
평균 지연 시간은 접촉군 (Exposed)의 한 개체가 병을 전염시킬 수 있게 되어 감염군으로 분류되기까지의 평균 시간을 나타내며 잠복기와는 다른데 이는 증상의 발현과 병을 전염시킬 수 있는 능력이 갖춰지는 것을 동치할 수 없기 때문이다. 감염률은 접촉률 (contact rate)로 불리기도 하는데, 더 상세하게는 로 는 감염 확률을, c는 접촉률을 나타낸다.
기초감염재생산수와의 관계
[편집]병의 확산이 멈추려면 와 의 조건이 성립해야 한다. 이 조건들을 방정식에 대입하면 와 가 되므로 라는 결과가 도출된다. 이는 기초감염재생산수 의 경우와 같고 이라고 하면[1][2]
가 된다. 일 때 정규화된 인명 수나 변형된 SEIR 공식을 사용하면 (SIR 모형에도 적용할 수 있으나 를 구하는 공식에서 의 정의가 다르다)
중간 단계로의 이행을 나타내는 매개변수 는 접촉군에 속한 환자가 사망할 가능성을 무시할 경우 기초감염재생산수에 영향을 주지 않는다.[1]
시간에 영향을 받는 순감염재생산수 (net reproduction number)는 일 때, 또는 로 정의할 수 있다.
최대 감염자 비율
[편집]기초감염재생산수는 개체군의 최대 감염자 비율에 큰 영향을 준다. 병에 감염된 개체 전체를 라고 하고 이면 위의 미분방정식에 의해
- ,
또는 기초감염재생산수 일 때
가 된다. 이 방정식을 로 바꾸고 변수분리법을 통한 적분에 의해 모든 t에 적용되는
가 도출되는데 여기서 은 자연로그를 뜻한다. 따라서
혹은
가 성립한다. 도움 함수 는 이므로 최대값이 이다. 최대 감염자 비율 에는 일 때 도달하게 되며, 따라서 는 기초감염재생산수와 초기값 및 만의 영향을 받는다:
아직 알려지지 않은 바이러스 등에 의해 새로이 등장하는 전염병의 경우 이고 으로 설정되고 최대 감염자 비율과 기초감염재생산수의 관계는 다음과 같아진다.
이 공식은 SIR 모형의 최대 감염자 비율을 구하는 공식과 일치한다.
전염병 유행 말기 회복된 개체의 비율
[편집]격리가 전혀 없이 전염병이 유행할 경우 개체군 내에서 감염된 전체 개체의 비율도 기초감염재생산수와 상관 관계가 있다. 미분방정식으로 나타낼 경우
이고 초기값이 라면
이다. 일 경우 이고 따라서 이 된다. 여기서
를 도출할 수 있고 다음과 같은 공식에 이르게 된다.
이고 라고 했을 때 최종적인 공식은 가 되고 람베르트 W 함수 를 활용해 로 바꾼 뒤 역치환하는 과정을 거쳐
에 도달하게 된다.
W 함수의 필요한 부분을 계산하려면 함수의 2차 테일러 다항식에 −1 을 넣고 영점을 구한 뒤 고정 소수점 반복을 로 한차례 적용한다. 그 결과물은
이다. W 함수의 가까운 근사치는
에 의해 얻을 수 있고 여기서 는 뉴턴 방법의 n 번째 반복인
을 뜻한다. 현실의 전염병에 적용할 때는 이면 충분하다.
기하급수적 초기 단계
[편집]전염병의 초기 단계에서는 확산이 기하급수적인 형태에 가깝게 일어난다. 을 가정한다면 역시 참이 된다. 그에 따라 와 이 성립하며 따라서
라는 결과가 나온다. 이 방정식을 양쪽 다 미분한 후 로 치환하고 로 나누면
혹은
를 얻게 된다. 초기에는 가 상당히 정확한 근삿값이므로 이 성립하고 매개변수 와 증식상수 의 관계를 알아낼 수 있다.
다른 체계적 접근법으로는 를 곧바로 1로 정의하고 미분방정식을 연립 일차 방정식으로 단순화하여
로 설명하는 것이 가능하다.[5] 다른 접근 방법들도 고정계수와 고유값&고유벡터를 활용한 선형 상미분방정식에 기반한다. 증식상수는 계수행렬 의 고유값으로 이미 발견한 방정식은
에 의해 구할 수 있다. 모든 기하급수적 증식과정에서와 마찬가지로 증식상수 는 더 이해가 쉽게
를 통해 수 배의 증식이 이루어지기까지의 시간으로 나타낼 수 있으며, 이는 특히 배가시간 에도 적용 가능하다.
예시
[편집]다음은 독일의 코로나19 범유행을 모델링할 때 쓰인 매개변수를 파이썬에서 그대로 적용한 예시이다.[6] 전염병의 확산은 기초감염재생산수 를 토대로 이루어지고, 이는 중요한 격리 조치들이 취해지지 않았을 경우에 해당한다. 기초값 문제를 수치적으로 푸는데는 오일러 방법이면 충분하다.
from numpy import array as vector
# Explicit Euler method
def euler_method(f,t0,x0,t1,h):
t = t0; x = x0
a = [[t,x]]
for k in range(0,1+int((t1-t0)/h)):
t = t0 + k*h
x = x + h*f(t,x)
a.append([t,x])
return a
def SEIR_model(beta,gamma,a):
def f(t,x):
S,E,I,R = x
return vector([
-beta*S*I,
beta*S*I - a*E,
a*E - gamma*I,
gamma*I
])
return f
def SEIR_simulation(beta,gamma,a,E0,I0,days,step=0.1):
x0 = vector([1.0-E0-I0,E0,I0,0.0])
return euler_method(SEIR_model(beta,gamma,a),0,x0,days,step)
def diagram(simulation):
import matplotlib.pyplot as plot
plot.style.use('fivethirtyeight')
figure,axes = plot.subplots()
figure.subplots_adjust(bottom = 0.15)
axes.grid(linestyle = ':', linewidth = 2.0, color = "#808080")
t,x = zip(*simulation())
S,E,I,R = zip(*x)
axes.plot(t,S, color = "#0000cc")
axes.plot(t,E, color = "#ffb000", linestyle = '--')
axes.plot(t,I, color = "#a00060")
axes.plot(t,R, color = "#008000", linestyle = '--')
plot.show()
def simulation1():
N = 83200000 # Einwohnerzahl von Deutschland 2019/2020
R0 = 2.4; gamma = 1/3.0
return SEIR_simulation(
beta = R0*gamma, gamma = gamma, a = 1/5.5,
E0 = 40000.0/N, I0 = 10000.0/N, days = 140)
diagram(simulation1)
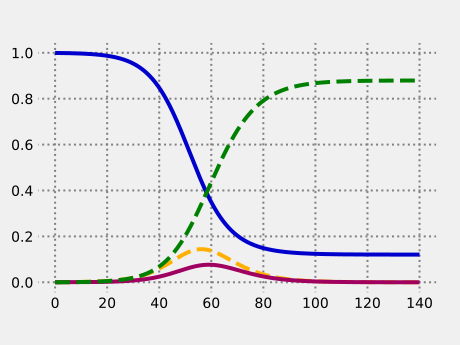
S, E, I, R의 네 군이 전체 개체군에서 차지하는 비율을 날짜별로 계산한 결과. S는 청색, E는 노란색 점선, I는 자홍색, R는 녹색 점선으로 표시. |
여기서 볼 수 있는 것은 전염병이 면역임계치 (critical immune threshold)
에 도달한 다음에도 감염군에 의해 계속 전파된다는 점이다. 다만 전염병이 면역임계치에 도달한 후에는 약 한달간의 강력한 격리 조치를 통해 멈추도록 만들 수 있을 것이다.
인구통계역학을 포함할 경우
[편집]
사망률 를 상수로 두고 이에 상응하는 출생률과 함께 모형을 확장시킨
공식도 있다. 원래 SEIR 모형과는 다르게 전염병의 풍토병화를 전제하며
라고 정의한 평형에 이르기까지 감염대상군 에서는 진동이 올 수도 있다.
기초감염재생산수는 여기서 평형 상태를 놓고 볼 때 다음과 같이 정의할 수 있다.
시간의존적 전염률 (Time-dependent transmission rate)
[편집]전염병을 대하는 태도의 변화, 격리와 계절성은 전염률의 변화를 가져오고, 이는 전염률을 시간의존적 함수 로 모델링할 때 고려되는 사안이다. 이 때에도 다른 모형들은 달라지지 않는다.[7] 계절성을 가장 단순하게 모델링하는 방법으로는 사인 진동 (sine oscillation) 형태로 날씨가 추운 달에는 전염률이 높고 따뜻한 달에는 전염률이 낮게 나오는 것을 가정할 수가 있다.[8]
명백한 시간의존도를 가진 미분방정식 체계는 자생적이지 않고 따라서 더 이상 직접적으로 역동적인 체계라고 할 수 없다. 다만 의 추가를 통해 인위적으로 자생적인 체계를 갖출 수는 있다.
외부 링크
[편집]- SEIR 모형 계산기 전염병의 경과를 SEIR 모형과 변수의 자유로운 입력을 통해 시뮬레이션하기
참고 문헌
[편집]- Matt J. Keeling, Pejman Rohani: Modeling Infectious Diseases in Humans and Animals. Princeton University Press, 2008.
- Matthias an der Heiden, Udo Buchholz: Modellierung von Beispielszenarien der SARS-CoV-2-Epidemie 2020 in Deutschland. Robert Koch-Institut, Abteilung für Infektionsepidemiologie (20. März 2020). DOI:10.25646/6571.2.
각주
[편집]- ↑ 가 나 Pauline van den Driessche, Reproduction numbers of infectious disease models, Infectious Disease Modelling, Band 2, August 2017, S. 288–303
- ↑ Odo Diekmann, Hans Heesterbeek, Tom Britton, Mathematical tools for understanding infectious disease dynamics, Princeton UP, 2013, S. 35
- ↑ Institute for Desease Modeling: SEIR Model Archived 2020년 5월 7일 - 웨이백 머신
- ↑ C. Hubbs (2020): Social Distancing to Slow the Coronavirus[깨진 링크(과거 내용 찾기)]
- ↑ Junling Ma: Estimating epidemic exponential growth rate and basic reproduction number. In: Infectious Disease Modelling, Volume 5, 2020, S. 129–141, KeAi Publishing (17. Dec. 2019).
- ↑ “Stellungnahme der Deutschen Gesellschaft für Epidemiologie (DGEpi) zur Verbreitung des neuen Coronavirus (SARS-CoV-2)” (PDF). 2020년 3월 20일에 원본 문서 (PDF)에서 보존된 문서. 2020년 6월 18일에 확인함. Die aktuelle Version siehe hier Archived 2020년 5월 29일 - 웨이백 머신.
- ↑ Gerardo Chowell, Cécile Viboud, Lone Simonsen, Seyed M. Moghadas: Characterizing the reproduction number of epidemics with early subexponential growth dynamics Archived 2020년 6월 19일 - 웨이백 머신. In: J. R. Soc. Interface 13: 20160659 (17. August 2016). DOI: 10.1098/rsif.2016.0659.
- ↑ M. Keeling, P. Rohani: Modeling Infectious Diseases in Humans and Animals. Abschnitt 5.2. (S. 159): Modeling forcing in childhood infectious diseases: Measles.





























































































