헬리케이스
헬리케이스 또는 헬리카제(Helicase)는 모든 생물에게서 필수적인 효소의 한 종류이다. 이들의 주기능은 유기체의 유전자를 포장 해제하는 것이다. 생물학적 시선으로 본다면 ATP를 가수분해하여 나온 에너지를 사용하여 2개의 결합된 핵산 가닥(즉, DNA, RNA, RNA-DNA 하이브리드)을 분리하여 핵산 인산다이에스터 결합의 방향을 따라 움직이는 모터 단백질이다. 가닥 분리를 위한 여러 공정이 있기 때문에 헬리케이스는 그 종류가 다양하다. 진핵 생물 유전자의 약 1%가 헬리케이스를 암호화한다.[1] 인간 게놈은 95개의 비중복 헬리케이스, 64개의 RNA 헬리케이스, 31개의 DNA 헬리케이스를 암호화한다.[2] DNA 복제, 전사, 번역, 재조합, DNA 수선, 리보솜 생합성과 같은 많은 세포 과정에서 헬리케이스의 사용을 필요로 하는 핵산 가닥의 분리를 포함한다.
기능[편집]
종종 헬리케이스는 ATP 가수분해로부터의 에너지를 사용하여 DNA 이중 나선 또는 자체 결합된 RNA 분자의 가닥을 분리하는데 사용되는데, 이 과정은 염기쌍 사이의 수소 결합을 끊는 것을 특징으로 한다. 또한 핵산 관련 단백질을 제거하고 상동 유전자 재조합을 촉진하는 기능을 한다.[3] 번역, 전사, 리보솜 생합성, RNA 스플라이싱, RNA 수송, RNA 편집, RNA 분해와 같은 RNA의 대사 과정은 모두 헬리케이스에 의해 촉진된다. 헬리케이스는 각각의 특정 효소에 특이적인 방향성 및 가공성을 갖는 이중체의 하나의 핵산 가닥을 따라 점진적으로 이동한다.
헬리케이스는 상이한 구조 및 올리고머화 상태를 채택한다. 반면 DnaB와 같은 헬리케이스는 반지 모양의 육각형으로서 DNA를 풀어주는 반면, 다른 효소들은 단량체나 이량체로 활동적인 것으로 나타났다. 연구 결과에 따르면 헬리케이스는 수동적으로 작용하여 촉매되지 않은 풀림이 일어나기를 기다린 다음 변위된 가닥 사이를 이동하거나[4], ATP 가수 분해에서 생성된 에너지를 사용하여 가닥 분리를 촉매하는 데 적극적인 역할을 수행할 수 있다.[5] 후자의 경우, 헬리케이스는 활성 모터와 유사하게 작용하여 ATP 가수 분해 효소 활성의 직접적인 결과로서 기판을 따라 풀고 이동한다.[6] 헬리케이스는 포크 접합의 불안정화를 돕는 보조 단백질의 존재로 인해 인 비트로보다 인 비보에서 훨씬 더 빠르게 처리될 수 있다.
헬리케이스 활동의 활성화 장벽[편집]

핵산을 제거하는 것과 같은 효소 헬리케이스 작용은 각 특정 작용[7]의 활성화 장벽()의 하강을 통해 달성된다. 활성화 장벽은 다양한 요인의 결과로서 다음과 같은 방정식을 사용하여 정의할 수 있다.
= 풀린 염기쌍의 수(bps)
= 염기쌍 형성의 자유 에너지
= 헬리케이스로 인한 자유 에너지의 감소
= 압축 해제력으로 인한 자유 에너지 감소[7]
활성화 장벽의 높이에 기여하는 요인으로는 관련 분자의 특정 핵산 배열, 관련된 염기쌍 수, 복제 포크의 장력, 불안정한 힘이 있다.[7]
능동적 및 수동적 헬리케이스[편집]
헬리케이스에 의해 극복되는 활성화 장벽의 크기는 능동적 또는 수동적 헬리케이스로부터 기여된다. 수동적 헬리케이스는 중요한 활성화 장벽이 존재한다(, 볼츠만 상수이고 시스템의 온도).[7] 이 중요한 활성 장애물 때문에, 그것의 풀림 진행은 크게 분자의 핵산 배열과 복제 포크에 작용하는 불안정한 힘의 존재에 의해 영향을 받는다. 특정 핵산 조합은 풀림 속도(구아닌 및 사이토신)를 감소시키는 반면, 다양한 불안정화 힘은 풀림 속도를 증가시킬 수 있다. 수동적 헬리케이스 시스템에서의 풀림 비율()이 이전 비율보다 적다.(, 단일 가닥 핵산, ssNA를 따른 전위). 또한 수동적 헬리케이스는 복제 포크에서 염기쌍의 일시적인 풀림에 의존하여 풀림 속도를 결정하는 것이다.
시스템에 상당한 장벽이 없는 능동적 헬리케이스에서는 헬리케이스가 핵산을 불안정하게 하여 핵산 순서에 관계없이 일정한 속도로 이중나선을 풀 수 있다().[7] 능동적 헬리케이스에서 와 는 대략 같다. 능동적 헬리케이스를 보는 또 다른 방법은 풀림을 촉진하기 위해 복제 포크를 직접 불안정하게 만드는 기능이다.
능동적 헬리케이스는 양측 가닥 핵산, dsNA 또는 ssNA 둘 다에서 작동 할 때 풀림 속도 및 전좌 속도와 관련하여 두 시스템 모두에서 유사한 행동을 나타낸다. 와 는 거의 같다.
또한 이 두 가지 범주의 헬리케이스는 메커니즘으로 모델링 될 수 있다. 이러한 모델에서 수동적 헬리케이스는 DNA 격자를 가로 지르는 열 변동 및 후속 이방성 구배에 의해 구동되는 브라운 래칫으로 개념화된다. 대조적으로, 활성 헬리 카제는 형태 적 "인치 웜"또는 핸드 오버 오버 "보행"메커니즘을 사용하여 스테핑 모터 (파워 스트로크 모터라고도 함)로 개념화된다.[8] 유기체에 따라, 이러한 나선 이송 과정은 5,000[9]에서 10,000[10] RPM의 회전 속도에서 발생할 수 있다.
DNA 헬리케이스의 역사[편집]
DNA 헬리케이스는 1976년 대장균에서 발견되었다. 이 헬리케이스는 '검출 분해없는 ATP 의존성 반응에서 DNA 이중체를 변성시키는 것으로 밝혀진 효소', 'DNA 풀림 효소'로서 기록되었다.[11] 최초의 진핵 생물 DNA 헬리케이스는 1978년 백합에서 발견되었다.[12] 그 이후로 DNA 헬리케이스는 다른 세균, 바이러스, 효모, 파리, 고등 진핵생물에서 발견되어 분리되어 왔다.[13] 현재까지 단일 세포 유기체, 박테리오파지의 6가지 헬리케이스, 바이러스의 12가지 헬리케이스, 효모의 15가지 헬리케이스, 식물의 8가지 헬리케이스, 종아리 흉선의 11가지 헬리케이스, 인간 세포의 25가지 이상의 헬리케이스가 분리되었다.[14] 다음은 헬리케이스 발견의 역사이다.
- 1976년 : 대장균에서 DNA 헬리케이스의 발견과 분리[11]
- 1978년 : 백합에서 분리된 최초의 진핵생물 DNA 헬리케이스 발견[12]
- 1982년 : 최초의 박테리오파지 DNA 헬리케이스(T4 gene 41 protein) 보고[13]
- 1985년 : 송아지 흉선으로부터 분리된 최초의 포유류 DNA 헬리케이스[15]
- 1986년 : SV40 큰 종양 항원은 바이러스의 헬리케이스로 보고됨[16]
- 1986년 : DNA 헬리케이스로 확인된 효모 단백질인 ATPase III 발견[17]
- 1988년 : 헬리케이스 모티프인 것으로 확인된 7개의 보존된 아미노산 도메인 발견
- 1989년 : DNA 헬리케이스 수퍼 패밀리 I과 수퍼 패밀리 II의 지정[18]
- 1989년 : DEAD box 헬리케이스 패밀리의 식별[19]
- 1990년 : 인간 DNA 헬리케이스의 분리[20]
- 1992년 : 최초 보고 된 미토콘드리아 DNA 헬리케이스의 분리[21]
- 1996년 : 완두콩에서 처음으로 정제된 엽록체 DNA 헬리케이스 발견 보고[22]
- 2002년 : 최초의 생화학 활성 말라리아 기생충(Plasmodium cynomolgi) DNA 헬리케이스의 분리[23]
구조적 특징[편집]
헬리케이스의 공통 기능은 이들이 어느 정도의 아미노산 서열 상동성을 나타내는 사실을 설명한다. 이들은 모두 1차 구조의 내부에 위치하며 핵산 기질을 따라 ATP 결합, ATP 가수분해 및 전위에 관여하는 서열 모티프를 갖는다. 아미노산 서열의 가변 부분은 각 헬리케이스의 특징과 관련이 있다.
이러한 헬리케이스 모티프의 존재는 추정 헬리케이스 활성이 주어진 단백질에 기인하는 것을 허용하지만, 반드시 활성 헬리케이스로서 이를 확인하는 것은 아니다. 그러나 보존된 모티프는 효소들 사이에서 진화적 상동성을 지지한다. 이러한 헬리케이스 모티프에 기초하여, 많은 헬리케이스 수퍼 패밀리가 구별되었다.
헬리케이스는 공유 서열 모티프에 기초하여 6개의 그룹(슈퍼 패밀리)으로 분류된다.[24] 반지 구조를 형성하지 않는 헬리케이스는 수퍼 패밀리 1, 2에 있고, 반지 구소 형성 헬리케이스는 수퍼 패밀리 3~6에 존재한다.[25] 또한 헬리케이스는 단일 또는 이중 가닥 DNA와 함께 작동하는지에 따라 α 또는 β로 분류된다. α 헬리케이스는 단일 가닥 DNA와 함께 작동하고, β 헬리케이스는 이중 가닥 DNA와 함께 작동한다. 또한 방향성에 따라 분류된다. 방향성이 3' 말단→5' 말단이면 A형이고, 방향성 5' 말단→3' 말단이면 B형이다.
- 수퍼 패밀리 1(SF1) : 이 수퍼 패밀리는 SF1A 및 SF1B 헬리케이스로 더 세분 될 수 있다.[24] 이 그룹에서 헬리케이스는 3' 말단→5' 말단(SF1A 서브 패밀리) 또는 5' 말단→3' 말단(SF1B 서브 패밀리)의 방향성을 가질 수 있다.[26] 가장 알려진 SF1A 헬리케이스는 그람 음성균에서 Rep, UvrD이고, 그람 양성균에서 PcrA 헬리케이스이다. SF1B 그룹에서 가장 잘 알려진 헬리케이스는 RecD, Dda이다.
- 수퍼 패밀리 2 (SF2) : 이것은 다양한 세포 과정에 관여하는 가장 큰 헬리케이스 그룹이다.[27] 이것들은 Q, I, Ia, Ib, II~ VI까지 총 9개의 보존된 모티프가 존재한다. 이 그룹은 주로 DEAD 상자 RNA 헬리케이스로 구성된다.[25] SF2에 포함된 다른 일부 헬리케이스는 RecQ 유사 패밀리와 Snf2 유사 효소이다. 대부분의 SF2 헬리케이스는 XPD군과 같은 몇 가지 예외를 제외하고 A형이다.
- 수퍼 패밀리 3 (SF3) : 수퍼 패밀리 3은 주로 작은 DNA 바이러스와 일부 큰 핵 세포질 DNA 바이러스에 의해 암호화 된 헬리케이스로 구성된다.[28][29] 이것들은 3' 말단→5' 말단의 방향성을 가지며, 이는 모두 A형 헬리케이스임을 의미한다. 가장 잘 알려진 SF3 헬리케이스는 유두종바이러스 E1 헬리케이스이다.
- 수퍼 패밀리 4 (SF4) : 모든 SF4 패밀리 헬리케이스는 B형이다(5' 말단→3' 말단). 가장 많이 연구 된 SF4 헬리케이스는 박테리오파지 T7의 gp4이다.
- 수퍼 패밀리 5 (SF5) : Rho 단백질은 SF5 그룹과 일치한다.
- 수퍼 패밀리 6 (SF6) : SF3 분류에 포함되지 않은 핵심 AAA+를 포함한다. SF6 그룹의 일부 단백질은 MCM, RuvB, RuvA, RuvC이다.
헬리케이스 장애 및 질병[편집]
ATRX 헬리케이스 돌연변이[편집]
ATRX 유전자는 염색질 개장, 유전자 조절, DNA 메틸화와 같은 기능을 담당하는 것으로 생각되는 SNF2 서브 그룹 패밀리의 ATPX (XH2 및 XNP라고도 함) ATP 의존성 헬리케이스를 암호화한다.[30][31][32][33] 이러한 기능은 세포자살 예방에 도움을 주어 대뇌 피질 크기 조절을 야기할뿐만 아니라 해마 및 대뇌 피질 구조의 생존에 기여하여 기억과 학습에 영향을 미친다. 이 헬리케이스는 주위 동원체 헤테로크로마틴의 X 크로모좀(Xq13.1-q21.1)에 위치하고 헤테로크로마틴 단백질 1과 결합한다. 연구에 따르면 ATRX는 rDNA 메틸화에 중요한 역할을 하며, 배아 발달에 필수적이다.[34] 돌연변이는 ATRX 단백질 전체에서 발견되었으며, 이들 중 90% 이상이 아연 손가락 및 헬리케이스 도메인에 위치한다.[35] ATRX의 돌연변이는 알파 지중해빈혈 X-염색체 연관 정신지체 증후군(ATR-X 증후군)을 초래할 수 있다.
ATRX에서 발견되는 다양한 유형의 돌연변이는 가장 일반적으로 단일 염기 미스센스 돌연변이뿐만 아니라 넌센스, 프레임 시프트 및 결실 돌연변이를 포함하여 ATR-X와 연관된 것으로 밝혀졌다.[33] ATR-X의 특징은 소두증, 골격 및 얼굴 이상, 정신 지체, 생식기 이상, 발작, 언어 사용 및 능력 제한을 포함한다.[30][36][37] ATR-X에서 보이는 표현형은 ATRX 유전자의 돌연변이가 알파 글로빈 유전자와 같은 유전자 발현의 하향 조절을 야기한다는 것을 시사한다. 다른 환자에서 ATR-X의 다양한 특성을 발현시키는 원인은 여전히 알려져 있지 않다.
XPD 헬리케이스 점 돌연변이[편집]
XPD (Xeroderma pigmentosum factor D, 단백질 ERCC2라고도 함)는 철-황 클러스터 도메인을 포함하는 5' 말단→3' 말단, 수퍼 패밀리 II, ATP 의존적 헬리케이스이다.[38][39] XPD 헬리케이스에서의 점 돌연변이는 코카인 증후군(CS) 및 털유황이상증(TTD)과 같은 노화 장애와 관련이 있는 것으로 나타났다.[40] 코카인 증후군과 털유황이상증은 자외선에 대한 감수성과 조기 노화에 관련된 발달 장애이며, 코카인 증후군은 출생부터 심각한 정신 지체를 나타낸다. 또한 XPD 헬리케이스 돌연변이는 자외선에 대한 감수성을 특징으로 하는 피부병 발병의 수십배 증가를 야기하는 장애인 색소성 건피증(XP)과도 연관되어 있다.
XPD는 세포에서 전사 및 복구 인자인 TFIIH 복합체의 필수 성분이다.[40][41][42][43][44] 이 복합체의 일부로서, DNA를 풀어서 뉴클레오타이드 절단 복구를 촉진한다. TFIIH는 태양에 의한 손상을 입은 DNA의 수선을 지원한다. 이 복합체를 형성하고 그 기능에 기여하는 XPD 헬리케이스에서의 돌연변이는 3가지 질병 모두에서 보이는 햇빛에 대한 민감성을 야기할뿐만 아니라 XP에서 보이는 암, 털유황이상증, 코카인 증후군에서 보이는 조기 노화의 위험 증가를 유발한다.
털유황이상증으로 이어지는 XPD 헬리케이스 변이는 단백질-단백질 상호 작용에 관여하는 다양한 위치에서 단백질 전체에서 발견된다.[40] 이 돌연변이는 돌연변이 지점에서 다른 단백질과의 안정화 상호 작용을 형성 할 수 없기 때문에 불안정한 단백질을 초래한다. 이것은 차례대로 전체 TFIIH 복합체를 불안정화시켜 세포의 전사 및 복구 메커니즘에 결함을 초래한다.
코카인 증후군으로 이어지는 XPD 헬리케이스 돌연변이는 XPD 내의 돌연변이의 결과일 수 있으며, 이는 단백질의 강성을 야기하고, 이어서 수리 모드에서 잠금으로 인해 수리 기능에서 전사 기능으로 전환 할 수 없게한다.[40] 이것은 헬리케이스가 전사를 위한 DNA 세그먼트를 절단하게 할 수 있다. 현재의 증거가 XPD 헬리케이스의 결함을 지적하여 코카인 증후군의 경우 단백질에서 유연성의 손실을 초래하지만,이 단백질 구조가 코카인 증후군에 설명된 증상을 어떻게 유발하는지는 여전히 불분명하다.
색소성 건피증에서 XPD 헬리케이스 돌연변이는 ATP 또는 DNA 결합 부위에 존재한다.[40] 이것은 전사를 용이하게 할 수 있는 구조적으로 기능적인 헬리케이스를 야기하지만, DNA를 풀거나 DNA 수선에서 그의 기능을 억제한다. 태양으로 인한 손상으로 인한 것과 같은 돌연변이를 복구하는 세포의 능력의 부족은 색소성 건피증 환자에서 높은 암 발생률의 원인이다.
RecQ 패밀리 돌연변이[편집]

RecQ 헬리케이스(3' 말단→5' 말단)는 게놈의 안정성을 유지하고 부적절한 재조합을 억제하는 데 도움이 되는 수퍼 패밀리 II 그룹의 헬리케이스에 속한다.[45][46] RecQ 패밀리 헬리케이스에서의 결핍 또는 돌연변이는 비정상적인 유전자 재조합 또는 DNA 복제를 나타내며, 이는 염색체 불안정성 및 증식에 대한 전반적인 감소된 능력을 초래한다. 상동 재조합을 조절하는 역할을 하는 RecQ 패밀리 헬리케이스 BLM, RECQL4, WRN의 돌연변이는 상 염색체 열성 질환 블룸증후군(BS), 로트문드-톰슨 증후군(RTS), 베르너 증후군(WS)을 초래하는 것으로 밝혀졌다.[47]
블룸증후군은 평균 발병 연령이 24세인 암 발병 경향이 특징이다.[46][48] 블룸증후군 환자의 세포는 자매 염색체(SCE)와 과도한 염색체 손상 사이에 높은 빈도의 상호 교환을 보인다.[49] BLM이 복제 포크에서 파괴된 DNA 복제를 구제하는 데 중요한 역할을 한다는 증거가 있다.
베르너 증후군(Werner syndrome)은 조기 동맥경화, 골다공증 및 기타 연령 관련 질병, 육종 발생, 심근경색, 암 등이 조기에 발생하는 증후군이다.[46][50] 베르너 증후군 환자의 세포는 염색체 파괴 및 전위와 염색체 성분의 큰 결실로 인한 생식 수명이 감소하여 게놈 불안정성을 유발한다.
로트문드-톰슨 증후군은 조기 노화, 피부 및 골격 이상, 발진, 다형피부증, 청소년 백내장 및 골육종이 특징이다.[46][51] 게놈 불안정성을 야기하는 염색체 재배열은 로트문드-톰슨 증후군 환자의 세포에서 발견된다.
RNA 헬리케이스[편집]

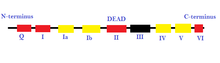
RNA 헬리케이스는 리보솜 생합성, pre-mRNA 스플라이싱 및 번역 개시와 같은 대부분의 RNA 대사 과정에 필수적이다. 또한 바이러스 RNA를 감지하는 데 중요한 역할을 한다.[52] RNA 헬리케이스는 척추 동물에서 외래 RNA를 확인할 수 있기 때문에 항 바이러스 면역 반응의 중재에 관여한다. 모든 바이러스의 약 80%는 RNA 바이러스이며 자체 RNA 헬리케이스를 포함한다.[53] 결함이 있는 RNA 헬리케이스는 암, 감염성 질환 및 신경 변성 장애와 관련이 있다. 결함이 있는 RNA 헬리케이스와 관련된 신경계 장애는 근위축성 측색 경화증, 척추성 근위축증, 척수 소뇌성 실조증 2형, 알츠하이머병이 있다.
RNA 헬리케이스와 DNA 헬리케이스는 SF6을 제외한 모든 헬리케이스 수퍼 패밀리에서 함께 발견되었다.[54][55] 최신으로 확인된 모든 진핵 생물의 RNA 헬리케이스는 비 고리 형태이고 SF1 및 SF2의 일부이다. 한편 고리 형태 RNA 헬리케이스는 세균 및 바이러스에서 발견되었다.[52] 그러나 모든 RNA 헬리케이스가 효소 기능, 즉 Swi / Snf 패밀리의 단백질에 의해 정의 된 바와 같은 헬리케이스의 활성을 나타내는 것은 아니다. 이들 단백질은 전형적인 헬리케이스 모티프를 보유하지만, ATP를 핵산 의존적 방식으로 가수 분해하고, 헬리케이스 코어 주위에 구축되며, 일반적으로 풀림 활성이 관찰되지 않는다.[56]
풀림 활성을 나타내는 RNA 헬리케이스는 적어도 2가지의 다른 메커니즘, 즉 표준 이중 풀림 및 국소 가닥 분리에 의해 특성화된다. 표준 이중 풀림은 DNA 풀림을 위해 이중가닥을 단계적으로 분리하는 것이다. 그러나 국소 가닥 분리는 헬리케이스가 이중체를 따라 임의의 위치에 로딩되는 공정에 의해 발생한다. 이것은 일반적으로 RNA의 단일 가닥 영역에 의해 도움이 되며, 효소의 로딩에는 ATP 결합이 필요하다.[57] 헬리케이스 및 ATP가 결합되면, 국소 가닥 분리가 발생하는데, 이는 ATP의 결합을 필요로 하지만 ATP 가수 분해의 실제 과정은 아니다.[58] 더 적은 염기쌍이 존재하면 이중 체는 효소의 도움없이 해리된다. 이 풀림은 DEAD/DEAH 상자 헬리케이스에 의해 사용된다.[59]
같이 보기[편집]
각주[편집]
- ↑ Wu Y (2012). “Unwinding and rewinding: double faces of helicase?”. 《J Nucleic Acids》 2012: 1–14. doi:10.1155/2012/140601. PMC 3409536. PMID 22888405.
- ↑ “Genome-wide comprehensive analysis of human helicases”. 《Commun Integr Biol》 4 (1): 118–37. January 2011. doi:10.4161/cib.13844. PMC 3073292. PMID 21509200.
- ↑ Patel, S. S.; Donmez, I (2006). “Mechanisms of Helicases”. 《Journal of Biological Chemistry》 281 (27): 18265–18268. doi:10.1074/jbc.R600008200. ISSN 0021-9258. PMID 16670085.
- ↑ “Real-time observation of bacteriophage T4 gp41 helicase reveals an unwinding mechanism”. 《PNAS》 104 (50): 19790–19795. 2007. Bibcode:2007PNAS..10419790L. doi:10.1073/pnas.0709793104. PMC 2148377. PMID 18077411.
- ↑ “Single-molecule studies reveal dynamics of DNA unwinding by the ring-shaped t7 helicase”. 《Cell》 129 (7): 1299–309. 2007. doi:10.1016/j.cell.2007.04.038. PMC 2699903. PMID 17604719.
- ↑ “Researchers solve mystery of how DNA strands separate”. 2007년 7월 3일. 2007년 7월 5일에 확인함.
- ↑ 가 나 다 라 마 “Active and passive mechanisms of helicases”. 《Nucleic Acids Res.》 38 (16): 5518–26. September 2010. doi:10.1093/nar/gkq273. PMC 2938219. PMID 20423906.
- ↑ Wu, C. G. and Spies, M.: Overview: What are Helicases? In: Spies, M. (Ed.): . Springer Science+Business Media, NY, 2013
- ↑ “Kevin Ahern's Biochemistry (BB 451/551) at Oregon State University”. 《oregonstate.edu》. 2021년 1월 26일에 원본 문서에서 보존된 문서. 2020년 3월 4일에 확인함.
- ↑ 3-D Animation Library; Replication: (Advanced)
- ↑ 가 나 “Enzymic unwinding of DNA. 2. Chain separation by an ATP-dependent DNA unwinding enzyme”. 《Eur. J. Biochem.》 65 (2): 441–9. June 1976. doi:10.1111/j.1432-1033.1976.tb10359.x. PMID 133023.
- ↑ 가 나 “DNA unwinding protein from meiotic cells of Lilium”. 《Biochemistry》 17 (10): 1872–80. May 1978. doi:10.1021/bi00603a011. PMID 207302.
- ↑ 가 나 “Bacteriophage T4 gene 41 protein, required for the synthesis of RNA primers, is also a DNA helicase.”. 《J. Biol. Chem.》 257 (20): 12426–34. October 1982. PMID 6288720.
- ↑ “Prokaryotic and eukaryotic DNA helicases. Essential molecular motor proteins for cellular machinery”. 《Eur. J. Biochem.》 271 (10): 1835–48. May 2004. doi:10.1111/j.1432-1033.2004.04093.x. PMID 15128294.
- ↑ “Mammalian DNA helicase”. 《Nucleic Acids Res》 13 (15): 5471–5483. 1985. doi:10.1093/nar/13.15.5471. PMC 321884. PMID 3162158.
- ↑ “DNA helicase activity of SV40 large tumor antigen”. 《EMBO J.》 5 (8): 1939–44. August 1986. doi:10.1002/j.1460-2075.1986.tb04447.x. PMC 1167061. PMID 3019672.
- ↑ “A new DNA-dependent ATPase which stimulates yeast DNA polymerase I and has DNA-unwinding activity”. 《J. Biol. Chem.》 261 (25): 11744–50. September 1986. PMID 3017945.
- ↑ “Two related superfamilies of putative helicases involved in replication, recombination, repair and expression of DNA and RNA genomes”. 《Nucleic Acids Res.》 17 (12): 4713–30. June 1989. doi:10.1093/nar/17.12.4713. PMC 318027. PMID 2546125.
- ↑ Linder, P., Lasko, P.F., Ashburner, M., Leroy, P., Nielson, P.J., Nishi, K., Schneir, J., Slonimski, P.P. (1989) Birth of the DEAD-box. Nature (London) 337, 121-122.
- ↑ “A DNA helicase from human cells”. 《Nucleic Acids Res.》 18 (23): 6785–92. December 1990. doi:10.1093/nar/18.23.6785. PMC 332732. PMID 1702201.
- ↑ “DNA helicase from mammalian mitochondria”. 《Proc. Natl. Acad. Sci. U.S.A.》 89 (18): 8562–6. September 1992. Bibcode:1992PNAS...89.8562H. doi:10.1073/pnas.89.18.8562. PMC 49960. PMID 1326759.
- ↑ “Purification and characterization of a DNA helicase from pea chloroplast that translocates in the 3'-to-5' direction”. 《Eur. J. Biochem.》 238 (1): 54–63. May 1996. doi:10.1111/j.1432-1033.1996.0054q.x. PMID 8665952.
- ↑ “Isolation and characterization of an eIF-4A homologue from Plasmodium cynomolgi”. 《Mol. Biochem. Parasitol.》 124 (1–2): 79–83. 2002. doi:10.1016/S0166-6851(02)00205-0. PMID 12387853.
- ↑ 가 나 Martin Singleton; Mark S. Dillingham; Dale B. Wigley (2007). “Structure and mechanism of Helicases and Nucleic Acid Translocases”. 《Annual Review of Biochemistry》 76: 23–50. doi:10.1146/annurev.biochem.76.052305.115300. PMID 17506634.
- ↑ 가 나 Margaret E. Fairman-Williams; Ulf-Peter Guenther; Echard Jankowsky (2010). “SF1 and SF2 helicases: family matters”. 《Current Opinion in Structural Biology》 20 (3): 313–324. doi:10.1016/j.sbi.2010.03.011. PMC 2916977. PMID 20456941.
- ↑ “Structural and Mechanistic Insight into DNA Unwinding by Deinococcus radiodurans UvrD”. 《PLoS ONE》 8 (10): :e77364. 2013. Bibcode:2013PLoSO...877364S. doi:10.1371/journal.pone.0077364. PMC 3797037. PMID 24143224.
- ↑ Pavan Umate; Narendra Tuteja; Renu Tuteja (2011). “Genome-wide comprehensive análisis of human helicases”. 《Communicative & Integrative Biology》 4 (1): 118–137. doi:10.4161/cib.13844. PMC 3073292. PMID 21509200.
- ↑ “Common origin of four diverse families of large eukaryotic DNA viruses”. 《J. Virol.》 75 (23): 11720–34. 2001. doi:10.1128/JVI.75.23.11720-11734.2001. PMC 114758. PMID 11689653.
- ↑ “Evolutionary history and higher order classification of AAA+ ATPases”. 《J. Struct. Biol.》 146 (1–2): 11–31. 2004. doi:10.1016/j.jsb.2003.10.010. PMID 15037234.
- ↑ 가 나 “X-linked mental retardation”. 《Nat. Rev. Genet.》 6 (1): 46–57. January 2005. doi:10.1038/nrg1501. PMID 15630421.
- ↑ Gibbons RJ, Picketts DJ, Villard L, Higgs DR (March 1995). “Mutations in a putative global transcriptional regulator cause X-linked mental retardation with alpha-thalassemia ATR-X syndrome”. 《Cell》 80 (6): 837–45. doi:10.1016/0092-8674(95)90287-2. PMID 7697714.
- ↑ Nextprot Online Protein Database. " ATRX-Transcriptional regulator ATRX.", Retrieved on 12 November 2012.
- ↑ 가 나 Picketts DJ, Higgs DR, Bachoo S, Blake DJ, Quarrell OW, Gibbons RJ (December 1996). “ATRX encodes a novel member of the SNF2 family of proteins: mutations point to a common mechanism underlying the ATR-X syndrome”. 《Hum. Mol. Genet.》 5 (12): 1899–907. doi:10.1093/hmg/5.12.1899. PMID 8968741.
- ↑ Gibbons R (2006). “Alpha thalassǢmia-mental retardation, X linked”. 《Orphanet J Rare Dis》 1: 15. doi:10.1186/1750-1172-1-15. PMC 1464382. PMID 16722615.
- ↑ “Alpha-Thalassemia X-Linked Intellectual Disability Syndrome”. 1993. PMID 20301622.
- ↑ Gibbons R (2006). “Alpha thalassaemia-mental retardation, X linked”. 《Orphanet J Rare Dis》 1: 15. doi:10.1186/1750-1172-1-15. PMC 1464382. PMID 16722615.
- ↑ “Mutations in a putative global transcriptional regulator cause X-linked mental retardation with alpha-thalassemia (ATR-X syndrome)”. 《Cell》 80 (6): 837–45. March 1995. doi:10.1016/0092-8674(95)90287-2. PMID 7697714.
- ↑ “Structure and mechanism of helicases and nucleic acid translocases”. 《Annu. Rev. Biochem.》 76: 23–50. 2007. doi:10.1146/annurev.biochem.76.052305.115300. PMID 17506634.
- ↑ “The helicase XPD unwinds bubble structures and is not stalled by DNA lesions removed by the nucleotide excision repair pathway”. 《Nucleic Acids Res.》 38 (3): 931–41. January 2010. doi:10.1093/nar/gkp1058. PMC 2817471. PMID 19933257.
- ↑ 가 나 다 라 마 “XPD helicase structures and activities: insights into the cancer and aging phenotypes from XPD mutations”. 《Cell》 133 (5): 789–800. May 2008. doi:10.1016/j.cell.2008.04.030. PMC 3055247. PMID 18510924.
- ↑ 《TFIIH enzymatic activities in transcription and nucleotide excision repair》. Methods in Enzymology 408. 2006. 246–63쪽. doi:10.1016/S0076-6879(06)08015-3. ISBN 9780121828134. PMID 16793373.
- ↑ Tirode F, Busso D, Coin F, Egly JM (January 1999). “Reconstitution of the transcription factor TFIIH: assignment of functions for the three enzymatic subunits, XPB, XPD, and cdk7”. 《Mol. Cell》 3 (1): 87–95. doi:10.1016/S1097-2765(00)80177-X. PMID 10024882.
- ↑ “Human xeroderma pigmentosum group D gene encodes a DNA helicase”. 《Nature》 365 (6449): 852–5. October 1993. Bibcode:1993Natur.365..852S. doi:10.1038/365852a0. PMID 8413672.
- ↑ “DNA repair helicase: a component of BTF2 (TFIIH) basic transcription factor”. 《Science》 260 (5104): 58–63. April 1993. Bibcode:1993Sci...260...58S. doi:10.1126/science.8465201. PMID 8465201.
- ↑ “Molecular genetics of RecQ helicase disorders”. 《Cell. Mol. Life Sci.》 64 (17): 2306–22. September 2007. doi:10.1007/s00018-007-7121-z. PMID 17571213.
- ↑ 가 나 다 라 “Junction of RecQ helicase biochemistry and human disease”. 《J. Biol. Chem.》 279 (18): 18099–102. April 2004. doi:10.1074/jbc.R300034200. PMID 15023996.
- ↑ “Homologous recombination and maintenance of genome integrity: cancer and aging through the prism of human RecQ helicases”. 《Mech. Ageing Dev.》 129 (7–8): 425–40. 2008. doi:10.1016/j.mad.2008.03.003. PMID 18430459.
- ↑ “The Bloom's syndrome gene product is homologous to RecQ helicases”. 《Cell》 83 (4): 655–66. November 1995. doi:10.1016/0092-8674(95)90105-1. PMID 7585968.
- ↑ “The Bloom's syndrome helicase (BLM) interacts physically and functionally with p12, the smallest subunit of human DNA polymerase delta”. 《Nucleic Acids Res.》 36 (16): 5166–79. September 2008. doi:10.1093/nar/gkn498. PMC 2532730. PMID 18682526.
- ↑ “The Werner syndrome protein is a DNA helicase”. 《Nat. Genet.》 17 (1): 100–3. September 1997. doi:10.1038/ng0997-100. PMID 9288107.
- ↑ “Mutations in RECQL4 cause a subset of cases of Rothmund-Thomson syndrome”. 《Nat. Genet.》 22 (1): 82–4. May 1999. doi:10.1038/8788. PMID 10319867.
- ↑ 가 나 Jankowsky, A.; Guenther, U. -P.; Jankowsky, E. (2010). “The RNA helicase database”. 《Nucleic Acids Research》 39 (Database issue): D338–D341. doi:10.1093/nar/gkq1002. PMC 3013637. PMID 21112871.
- ↑ Jankowsky E, 편집. (2010). 〈An introduction to RNA helicases: superfamilies, families, and major themes〉. 《RNA Helicases (RSC Biomolecular Sciences)》. Cambridge, England: Royal Society of Chemistry. 5쪽. ISBN 978-1-84755-914-2.
- ↑ Ranji, A.; Boris-Lawrie, K. (2010). “RNA helicases: Emerging roles in viral replication and the host innate response”. 《RNA Biology》 7 (6): 775–787. doi:10.4161/rna.7.6.14249. PMC 3073335. PMID 21173576.
- ↑ Jankowsky E (January 2011). “RNA helicases at work: binding and rearranging”. 《Trends Biochem. Sci.》 36 (1): 19–29. doi:10.1016/j.tibs.2010.07.008. PMC 3017212. PMID 20813532.
- ↑ “DEAD-box proteins unwind duplexes by local strand separation”. 《Mol. Cell》 28 (2): 253–63. October 2007. doi:10.1016/j.molcel.2007.08.016. PMID 17964264.
- ↑ “ATP hydrolysis is required for DEAD-box protein recycling but not for duplex unwinding”. 《Proc. Natl. Acad. Sci. U.S.A.》 105 (51): 20209–14. December 2008. Bibcode:2008PNAS..10520209L. doi:10.1073/pnas.0811115106. PMC 2629341. PMID 19088201.
- ↑ “DEAD-box proteins as RNA helicases and chaperones”. 《Wiley Interdiscip Rev RNA》 2 (1): 135–52. 2011. doi:10.1002/wrna.50. PMC 3032546. PMID 21297876.












