운반-전령 RNA
운반-전령 RNA(Transfer-Messenger RNA 또는 tmRNA)는 운반 RNA와 전령 RNA의 특성을 갖는 세균 RNA 분자이다. 운반-전령 RNA는 작은 단백질 B(SmpB), 신장 인자 Tu(EF-Tu), 리보솜 단백질 S1과 함께 리보 핵 단백질 복합체(tmRNP)를 형성한다. 트랜스 번역에서 운반-전령 RNA와 관련 단백질은 단백질 생합성 중간에 정지된 박테리아 리보솜에 결합한다. 예를 들어 정지 코돈이 존재하지 않는 RNA의 끝에 전령 RNA가 도달한 경우이다. 운반-전령 RNA는 정지된 리보솜을 재순환시키고, 완성되지 않은 폴리펩타이드에 단백질 분해를 유도하는 태그를 추가하여 비정상 전령 RNA의 분해를 촉진하는 역할을 한다.[1] 대부분의 세균에서 이러한 기능은 표준 일체형 운반-전령 RNA에 의해 수행된다.
발견과 초기 작업[편집]
대장균 RNA의 혼합된 10S 전기 영동 분획을 운반-전령 RNA 및 유사한 크기의 RNA 가수분해효소 P(10Sb)로 추가 분해 한 후, 운반-전령 RNA를 먼저 10Sa RNA로 지정하였다.[2] 혼합된 10S RNA에서 슈도유리딘의 존재는 운반-전령 RNA가 운반 RNA에서도 발견된 염기를 변형 시켰다는 것을 암시했다. 운반-전령 RNA의 3' 말단에서의 운반 RNA의 T 줄기-루프와의 유사성은 결핵균으로부터 ssrA를 시퀀싱 할 때 처음으로 인식되었다.[3] 후속 서열 비교는 알라닌-운반 RNA 연결효소에 의한 아미노아실화를 촉진하는 알라닌 운반 RNA의 것과 같은 요소를 갖는 수용체 줄기를 포함하여 운반-전령 RNA의 5' 말단과 3' 말단에 의해 형성된 완전한 운반 RNA 유사 도메인(TLD)을 나타냈다.[4] 또한 운반 RNA와의 차이점을 밝혀냈다. 안티코돈 암(Arm)은 운반-전령 RNA에서 누락되었고, D 암(Arm) 영역은 염기쌍이 없는 루프이다.
구조[편집]
표준 일체형 운반-전령 RNA의 2차 구조[편집]
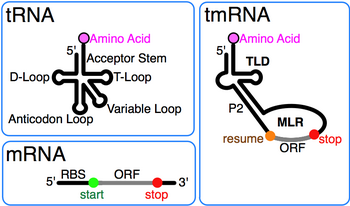
대장균 운반-전령 RNA의 2차 구조는 비교 서열 분석 및 구조적 조사에 의해 설명되었다.[5][6] 왓슨-크릭과 G-U 염기쌍은 수동 정렬 절차와 함께 자동화 된 계산 방법을 사용하여 세균의 운반-전령 RNA 서열을 비교함으로써 확인되었다.[7][8] 첨부된 그림은 프로토타입 운반-전령 RNA의 기본 결합 패턴을 보여준다. 이 운반-전령 RNA는 12개의 계통 발생적으로 지지된 나선(P1~P12라고도 함)으로 구성되며 일부는 나선 구조물로 나뉜다.
모든 운반-전령 RNA의 두드러진 특징은 보존된 운반 RNA 유사 도메인(TLD)이며, 나선 1, 12, 2a(각각 운반 RNA 수용체 및 수용체 줄기, T 줄기, 가변 줄기)로 구성되며 5' 일인산염을 포함한다. 그리고 분해 가능한 3' CCA가 종료된다. 전령 RNA 유사 영역(MLR)은 표준 운반-전령 RNA에서 유사 낫을 포함하는 큰 루프 및 태그 펩타이드에 대한 암호화 서열(CDS)이며, 계속 코돈 및 정지 코돈으로 표시된다. 암호화 된 태그 펩타이드(E. coli의 ANDENYALAA)는 이용가능한 단백질 가수분해효소 및 어댑터 세트에 따라 세균마다 다양하다.[9]
운반-전령 RNA는 전형적으로 4개의 유사 노트; 하나는 태그 펩타이드 CDS의 업스트림(pk1) 및 다른 3개의 유사 노트(pk2~pk4)는 CDS 다운 스트림을 포함한다. 일반적으로 의사 노트 영역은 보존되어 있지만 진화적으로 가변적이다. 예를 들어 남세균의 운반-전령 RNA에서, pk4는 2개의 탠덤 배열된 작은 유사 노트로 대체된다. 이것은 TLD 외부로 접히는 운반-전령 RNA가 중요 할 수 있음을 시사하지만, pseudoknot 영역에는 보존된 잔기가없고 psudoknots는 ssrA 서열이 plastid 및 endosymbiont 계통으로 분기됨에 따라 손실되는 첫 번째 구조 중 하나이다. 대장균의 운반-전령 RNA의 3개의 pseudoknot 영역의 염기쌍이 trans-translation 동안 중단된다.[7][10]
투피스(Two-Piece) 운반-전령 RNA[편집]
순환적으로 치환된 ssrA는 3가지 주요 계통으로 보고되었다.
- 모든 알파프로테오박테리아와 자코바 원생생물의 원시 미토콘드리아
- 2개의 분리 된 남세균 그룹(Gloeobacter, Prochlorococcus, Synechococcus를 포함하는 단계통군)
- 베타프로테오박테리아의 일부 구성원(Cupriavidus 와 Rhodocyclales)[11][12]
알파프로테오박테리아의 두 시퀀스(GGCRGUA 가진 전형적인 T 루프 시퀀스 TΨCRANY과 3' 말단의 슈도 대형 루프 시퀀스를 AACAGAA)가 미토콘드리아에서 MLR은 소실되었으며, 미토콘드리아 ssrA의 현저한 재순환은 Jakoba libera에서 작은 일체 생산물을 초래한다.[13]
남세균은 상이한 Synechococcus 균주에서 발생하는 두 유전자 유형 사이의 현저한 서열 유사성으로 인해 표준 유전자로부터 치환된 유전자의 진화에 대한 가장 타당한 사례를 제공한다.
운반-전령 RNA의 생산 과정[편집]
대부분의 운반-전령 RNA는 운반 RNA와 매우 유사하게 처리되는 더 큰 전구체로 전사된다. 5' 말단에서의 절단은 리보핵산가수분해효소 P가 담당한다.[4] 리보핵산분해효소 T와 리보핵산분해효소 PH가 가장 효과적이지만, 다수의 핵산외부가수분해효소가 운반-전령 RNA의 3' 말단의 처리에 참여할 수 있다.[14][15] 세균 종에 따라 3'-CCA는 운반 RNA 뉴클레오티딜 전달효소에 의해 암호화되거나 첨가된다.
치환된 전구체 운반-전령 RNA의 내부 부위에서의 유사한 처리는 물리적 분할이 두 조각으로 분리되는 것을 설명한다. 투피스 운반-전령 RNA에는 처리를 고려해야 하는 2개의 추가 말단이 있다. 알파프로테오박테리아의 경우, 하나의 5' 말단은 처리되지 않은 전사의 시작 부위이다.[16] 반면 3' 말단은 경우에 따라 rho 독립적 종료의 결과 일 수 있다.
3차원 입체 구조[편집]
완전한 운반-전령 RNA 분자의 고해상도 구조는 볼 수 없으며 MLR의 특유의 유연성으로 인해 획득하기 어렵다. 2007년, SmpB 단백질에 결합된 Thermus thermophilus TLD의 결정 구조는 3Å 분해능으로 얻어졌다. 이 구조는 SmpB가 표준 운반 RNA의 D 줄기 및 안티코돈을 모방하는 반면, 운반-전령 RNA의 나선형 부위 2a는 운반 RNA의 가변 암(Arm)에 상응한다는 것을 보여준다.[17] 트랜스 번역의 초기 단계에서 운반-전령 RNA의 저온전자현미경 연구는 리보솜과 tmRNP(운반-전령 RNA에 EF-tu, S1, SmpB와 같은 단백질이 붙은 복합체의 상태) 사이의 공간적 관계를 보여준다. TLD는 50S 리보솜 서브 유닛에서 GTP 가수분해효소 연관 센터 근처에 위치하고 나선 5 및 유사 노트(pk2~pk4)는 30S 리보솜 서브 유닛의 주위에 호를 형성한다.[18]


트랜스 번역[편집]

운반-전령 RNA에 의한 암호화는 1995년에 발견하였다.[20] Simpson과 동료들이 대장균에서 쥐 사이토카인 인터루킨 6을 과발현하고 각각 동일한 11개의 아미노산 잔기 연장으로 카복실기 말단에 태그된 다수의 절단된 사이토카인 유래 펩타이드를 발견했다 (AANDENYALAA. 운반-전령 RNA 자체의 3' 말단에서 오는 N 말단 알라닌을 제외하고, 이 태그 서열은 대장균 운반-전령 RNA에서 짧은 개방형 판독 프레임으로 추적되었다. 태그 펩타이드 수여하고 그 인식 단백질을 분해하는, 운반-전령 RNA 조치에 대한 트랜스 번역 모델이 제안되었다.[21]
트랜스 번역 메커니즘에 대한 자세한 내용은 현재 연구중이지만, 운반-전령 RNA가 먼저 정지 된 리보솜의 빈 A자리를 차지한다는 것은 밝혀졌다. 후속적으로 리보솜은 절단된 전령 RNA의 3' 말단에서 MLR의 계속 코돈으로 이동한 다음, 인 프레임 운반-전령 RNA 종결 코돈이 발생할 때까지 번역이 정상적으로 계속되는 단계가 이어진다. 일부 세균 종에서는 트랜스 번역이 필수적이지만, 다른 세균종에서는 스트레스가 많은 성장 조건에서 생존하기 위해서는 운반-전령 RNA가 필요하다.[22] 유기체에 따라, 태그 펩타이드는 다양한 단백질 가수 분해 효소 또는 단백질 가수 분해 효소 어댑터에 의해 인식된다.[9]
미토콘드리아 운반-전령 RNA (ssrA 유전자)[편집]
미토콘드리아 운반-전령 RNA(mt-tmRNA)는 미토콘드리아에서 암호화 된 운반-전령 RNA로 레크리노모나스속에 의해 가정되었다.[11] 또한 운반-전령 RNA를 암호화하는 미토콘드리아 유전자(ssrA)의 존재 및 전사, RNA 처리 부위가 자코바류의 한 구성원을 제외한 모든 구성원에 대해 확인되었다.[23] 알라닌을 이용한 미토콘드리아 운반-전령 RNA 아미노아실화는 Jakoba libera에 이용가능하다.[13] 최근 ssrA도의 난균류의 미토콘드리아 게놈에서 확인되었다.[24] 알파프로테오박테리아(미토콘드리아의 조상)에서와 같이, 미토콘드리아 운반-전령 RNA는 유전자가 원피스 운반-전령 RNA 입체 형태를 암호화하는 것으로 되돌아 간 Jakoba libera를 제외하고는 원형 치환된 투피스 RNA 분자이다.
미토콘드리아 게놈에서 ssrA의 식별[편집]

미토콘드리아 운반-전령 RNA 유전자는 초기에 자코바류 사이에 보존되고 별개의 운반 RNA 유사 2차 구조로 접힐 가능성이 있는 짧은 서열로 인식되었다.
미토콘드리아 운반-전령 RNA의 구조[편집]
세균 운반-전령 RNA는 운반 RNA (Ala) 유사 도메인(종결 코돈이 결여된 전령 RNA에 비암호화 된 알라닌의 첨가를 허용함), 폴리펩타이드를 대상으로하는 단백질 태그를 암호화하는 전령 RNA 유사 도메인으로 구성된다. 전령 RNA 유사 도메인은 미토콘드리아 운반-전령 RNA에서 손실되었다. 비교 서열 분석은 미토콘드리아 운반-전령 RNA에 전형적인 특징을 나타낸다.[24] 가장 잘 보존된 것은 아미노아실 수용체 줄기의 1차 서열이다. 이 부분은 판별자 위치에서 변하지 않는 A 잔기 및 위치 3에서 GU 쌍을 갖는다(GC 쌍을 갖는 Seculamonas ecuadoriensis는 제외). 이 위치는 알라닐 운반 RNA 합성 효소에 대한 인식 부위이다. P2는 가변 길이(3~10개의 염기쌍)의 나선이고 운반 RNA의 안티코돈 줄기에 상응하지만, 안티코돈 루프는 없다(운반-전령 RNA 기능에는 필요하지 않음). P2는 운반 RNA 유사 구조를 안정화시키지만, 동위 원소에 걸쳐 변하지 않는 4개의 뉴클레오타이드가 추가의 현재 미확인 기능을 시사한다. P3은 5개의 염기쌍을 갖고 운반 RNA의 T 암(Arm)에 상응하지만, 짝 지어진 영역 및 루프 모두에서 상이한 공통 뉴클레오타이드를 갖는다. T 루프 서열간에 보존되어 난균류 및 자코바류에 단지 약간의 편차가 있을 뿐이다. 마지막으로 세균 운반-전령 RNA에 대한 단축된 3 뉴클레오타이드 D 루프 특성을 갖는 운반 RNA 유사 D 줄기 대신, 미토콘드리아 운반-전령 RNA는 매우 가변적인 5~14 뉴클레오타이드의 긴 루프를 갖는다. 투피스 미토콘드리아 운반-전령 RNA의 계속 서열(Int.)은 A+U 풍부하고 불규칙한 길이(4~34 뉴클레오타이드)이다.). 원피스 및 투피스 미토콘드리아 운반-전령 RNA의 2차 구조 모델은 그림 1을 참조하라.
미토콘드리아 운반-전령 RNA 처리 및 발현[편집]

Phytophthora sojae의 RNA-Seq 데이터는 이웃하는 미토콘드리아 운반 RNA와 유사한 발현 수준을 보여 주며, 4개의 주요 가공 장소는 성숙한 미토콘드리아 운반-전령 RNA의 예측된 말단을 보여준다.[24] 운반-전령 RNA 전구체 분자는 리보 핵산 가수 분해 효소 P 및 운반 RNA 3' 말단 가공 핵산 중간 분해 효소에 의해 처리될 수 있다(그림 2 참조).
같이 보기[편집]
참고[편집]
각주[편집]
- ↑ “Biology of trans-translation”. 《Annual Review of Microbiology》 62: 133–51. 2008. doi:10.1146/annurev.micro.62.081307.162948. PMID 18557701.
- ↑ “Characterization of 10S RNA: a new stable rna molecule from Escherichia coli”. 《Molecular & General Genetics》 174 (1): 25–32. July 1979. doi:10.1007/BF00433301. PMID 384159.
- ↑ “Identification of the 10Sa RNA structural gene of Mycobacterium tuberculosis”. 《Nucleic Acids Research》 20 (1): 138. January 1992. doi:10.1093/nar/20.1.138. PMC 310338. PMID 1371186.
- ↑ 가 나 “A tRNA-like structure is present in 10Sa RNA, a small stable RNA from Escherichia coli”. 《Proceedings of the National Academy of Sciences of the United States of America》 91 (20): 9223–7. September 1994. doi:10.1073/pnas.91.20.9223. PMC 44784. PMID 7524073.
- ↑ “Phylogenetic analysis of tmRNA secondary structure”. 《RNA》 2 (12): 1306–10. December 1996. PMC 1369456. PMID 8972778.
- ↑ “Probing the structure of the Escherichia coli 10Sa RNA (tmRNA)”. 《RNA》 3 (1): 89–103. January 1997. PMC 1369465. PMID 8990402.
- ↑ 가 나 “Comparative sequence analysis of tmRNA”. 《Nucleic Acids Research》 27 (10): 2063–71. May 1999. doi:10.1093/nar/27.10.2063. PMC 148424. PMID 10219077.
- ↑ “Semiautomated improvement of RNA alignments”. 《RNA》 13 (11): 1850–9. November 2007. doi:10.1261/rna.215407. PMC 2040093. PMID 17804647.
- ↑ 가 나 “Evolution of the ssrA degradation tag in Mycoplasma: specificity switch to a different protease”. 《Proceedings of the National Academy of Sciences of the United States of America》 105 (42): 16113–8. October 2008. doi:10.1073/pnas.0808802105. PMC 2570983. PMID 18852454.
- ↑ “Transfer-messenger RNA unfolds as it transits the ribosome”. 《RNA》 11 (5): 668–73. May 2005. doi:10.1261/rna.7269305. PMC 1370753. PMID 15811920.
- ↑ 가 나 “tmRNAs that encode proteolysis-inducing tags are found in all known bacterial genomes: A two-piece tmRNA functions in Caulobacter”. 《Proceedings of the National Academy of Sciences of the United States of America》 97 (14): 7778–83. July 2000. doi:10.1073/pnas.97.14.7778. PMC 16621. PMID 10884408.
- ↑ “A third lineage with two-piece tmRNA”. 《Nucleic Acids Research》 32 (15): 4531–8. 2004. doi:10.1093/nar/gkh795. PMC 516066. PMID 15326226.
- ↑ 가 나 “Loss of the mRNA-like region in mitochondrial tmRNAs of jakobids”. 《RNA》 10 (4): 605–14. April 2004. doi:10.1261/rna.5227904. PMC 1370551. PMID 15037770.
- ↑ “Characterization of the RNA processing enzyme RNase III from wild type and overexpressing Escherichia coli cells in processing natural RNA substrates”. 《The International Journal of Biochemistry》 24 (5): 737–49. May 1992. doi:10.1016/0020-711X(92)90007-N. PMID 1375563.
- ↑ “3' exoribonucleolytic trimming is a common feature of the maturation of small, stable RNAs in Escherichia coli”. 《Proceedings of the National Academy of Sciences of the United States of America》 95 (6): 2856–61. March 1998. doi:10.1073/pnas.95.6.2856. PMC 19659. PMID 9501180.
- ↑ “Variations on the tmRNA gene”. 《RNA Biology》 6 (4): 355–61. 2009. doi:10.4161/rna.6.4.9172. PMID 19617710.
- ↑ 가 나 “Structural basis for functional mimicry of long-variable-arm tRNA by transfer-messenger RNA”. 《Proceedings of the National Academy of Sciences of the United States of America》 104 (20): 8293–8. May 2007. doi:10.1073/pnas.0700402104. PMC 1895943. PMID 17488812.
- ↑ “Visualizing tmRNA entry into a stalled ribosome”. 《Science》 300 (5616): 127–30. April 2003. doi:10.1126/science.1081798. PMID 12677067.
- ↑ “Solution structure of a tmRNA-binding protein, SmpB, from Thermus thermophilus”. 《FEBS Letters》 535 (1–3): 94–100. January 2003. doi:10.1016/S0014-5793(02)03880-2. PMID 12560085.
- ↑ “C-terminal extension of truncated recombinant proteins in Escherichia coli with a 10Sa RNA decapeptide”. 《The Journal of Biological Chemistry》 270 (16): 9322–6. April 1995. doi:10.1074/jbc.270.16.9322. PMID 7536743.
- ↑ “Role of a peptide tagging system in degradation of proteins synthesized from damaged messenger RNA”. 《Science》 271 (5251): 990–3. February 1996. doi:10.1126/science.271.5251.990. PMID 8584937.
- ↑ Ahmed, Niyaz, 편집. (2008). “Trans-translation in Helicobacter pylori: essentiality of ribosome rescue and requirement of protein tagging for stress resistance and competence”. 《PLOS ONE》 3 (11): e3810. doi:10.1371/journal.pone.0003810. PMC 2584231. PMID 19043582.
- ↑ “Strikingly bacteria-like and gene-rich mitochondrial genomes throughout jakobid protists”. 《Genome Biology and Evolution》 5 (2): 418–38. 2013. doi:10.1093/gbe/evt008. PMC 3590771. PMID 23335123.
- ↑ 가 나 다 “A second eukaryotic group with mitochondrion-encoded tmRNA: in silico identification and experimental confirmation”. 《RNA Biology》 10 (7): 1117–24. July 2013. doi:10.4161/rna.25376. PMC 3849159. PMID 23823571. 2014년 2월 21일에 원본 문서에서 보존된 문서. 2014년 2월 13일에 확인함.
추가 자료[편집]
- “Cell cycle-regulated degradation of tmRNA is controlled by RNase R and SmpB”. 《Molecular Microbiology》 57 (2): 565–75. July 2005. doi:10.1111/j.1365-2958.2005.04709.x. PMC 3776457. PMID 15978085.
