다이서
엔도리보뉴클레이스 효소 다이서(Dicer)는[1] 인간에서 다이서1 유전자에 의해 암호화되는 효소이다. RNase III 그룹 제품군의 일부인 다이서는 이중가닥 RNA (dsRNA) 및 pre-microRNA(pre-miRNA)를 각각 small interfering RNA 및 microRNA라고 하는 짧은 이중가닥 RNA 단편으로 절단한다. 이 단편은 길이가 약 20 – 25개 염기쌍이고 3'-말단에 2개 염기 돌출부가 있다. 다이서는 RNA 간섭에 필수적인 RNA 유도 침묵 복합체(RISC)의 활성화를 촉진한다. RISC에는 전령 RNA (mRNA)를 분해할 수 있는 엔도뉴클레아 제인 촉매 성분인 Argonaute가 있다.
RNAi(RNA 간섭현상)는 유전자의 발현을 조절하는 기작으로, 세포 안에 생성되거나 외부에서 유입된 이중 나선 RNA (dsRNA)나, miRNA 전구체가 siRNA나 miRNA로 만들어지고, 이렇게 만들어진 siRNA, miRNA 가 유전자의 전사를 조절하거나, mRNA를 분해 또는 단백질의 전사를 제어하여 유전자의 발현을 조절하는 기작이다.[1]
발견[편집]
다이서라는 이름은 2001년에 Stony Brook 박사 과정 학생인 Emily Bernstein이 Cold Spring Harbor Laboratory의 Gregory Hannon의 연구실에서 연구를 수행하던 중에 주어졌다. 번스타인은 이중가닥 RNA에서 작은 RNA 단편을 생성하는 효소를 발견하려고 했다. 약 22개의 뉴클레오티드 RNA 단편을 생성하는 다이서의 능력은 dsRNA 형질감염으로 RNAi 경로를 시작한 후 RISC 효소 복합체에서 분리함으로써 발견되었다. 이 실험은 RISC가 관찰 가능한 작은 뉴클레오티드 단편을 생성하는 데 책임이 없다는 것을 보여주었다. RNA 단편을 생성하는 RNase III 그룹 계열 효소의 능력을 테스트하는 후속 실험은 현재 다이서로 명명된 Drosophila CG4792로 검색 범위를 좁혔다.
다이서 orthologs는 다른 많은 유기체에 존재한다.[2] 이끼 Physcomitrella patens DCL1b는 4개의 다이서 단백질 중 하나이며 miRNA 생합성에 관여하지 않고 miRNA 표적 전사체를 절단하는 데 관여한다. 따라서, 유전자 발현 조절을 위한 새로운 메커니즘, 즉 miRNA에 의한 유전자의 후성 유전학적 침묵이 발견되었다.[3]
결정 구조의 관점에서, 탐색된 첫 번째 다이서는 원생동물 Giardia Internalis의 다이서였다. 이 작업은 버클리 캘리포니아 대학의 Jennifer Doudna 연구실에서 박사후 연구원으로 연구를 수행하는 동안 Ian MacRae가 수행했다. PAZ 도메인과 2개의 RNase III 그룹이 X선 결정학에 의해 발견되었다. 단백질 크기는 82이다(이 크기가 무슨 뜻인지 대략 짐작하려면, 예를 들어 다른 유기체의 더 큰 다이서 단백질에서 후속적으로 발견된 보존된 기능적 코어를 나타내는 kDa는 219이다). 인간의 kDa. 인간과 G. Internalis 다이서의 크기 차이는 인간 다이서 내에 최소 5개의 서로 다른 도메인이 존재하기 때문이다. 이들 도메인은 다이서 활성 조절, dsRNA 처리 및 RNA 간섭 단백질 인자 기능에 중요하다.[4]
기능[편집]

인간 다이서(hs다이서 또는 다이서1이라고도 함)는 헬리카제와 PAZ(Piwi / Argonaute /Zwille) 도메인을 모두 포함하기 때문에 리보뉴클레이스 효소 III 그룹으로 분류된다.[6][7] 이러한 도메인 외에도 hs다이서에는 2개의 RNaseIII 그룹과 2개의 이중가닥 RNA 결합 도메인(DUF283 및 dsRBD)과 같은 4개의 다른 기능적 도메인이 포함되어 있다.[4][8]
현재 연구에 따르면 PAZ 도메인은 dsRNA의 2개 뉴클레오타이드 3' 돌출부에 결합할 수 있는 반면 RNaseIII 그룹 촉매 도메인은 가닥 절단을 시작하기 위해 dsRNA 주위에 유사-이량체를 형성한다. 이것은 dsRNA 가닥의 기능적 단축을 초래한다. PAZ와 RNaseIII 그룹 사이의 거리는 커넥터 나선의 각도에 의해 결정되며 마이크로 RNA 제품의 길이에 영향을 준다.[5] dsRBD 도메인은 dsRNA에 결합하지만 도메인의 특정 결합 부위는 정의되지 않았다. 이 도메인은 RNaseIII 그룹을 효과적으로 배치하여 sRNA 제품의 특이성을 제어하기 위해 다른 조절 단백질 (인간의 TRBP, R2D2, 초파리의 Loqs)과의 복합체의 일부로 작동할 수 있다.[9] 헬리카제 도메인은 긴 기질을 처리하는 것과 관련이 있다.[9]
RNA 간섭에서의 역할[편집]
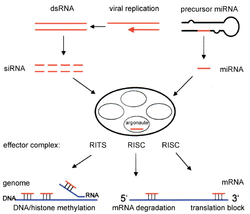
마이크로 RNA[편집]
RNA 간섭은 RNA 분자가 miRNA로 분해되어 특정 숙주 mRNA 서열의 유전자 발현을 억제하는 과정이다. miRNA는 핵의 1차 miRNA(pri-miRNA)에서 시작하여 세포 내에서 생성된다. 이러한 긴 서열은 더 작은 전구체 miRNA(pre-miRNA)로 절단되며, 이는 일반적으로 헤어핀 구조를 갖는 70개 뉴클레오티드이다. Pri-miRNA는 DGCR8에 의해 식별되고 Drosha에 의해 절단되어 핵에서 발생하는 과정인 pre-miRNA를 형성한다. 이 pre-miRNA는 세포질로 내보내지고 다이서에 의해 절단되어 성숙한 miRNA를 형성한다.[11]
작은 간섭 RNA[편집]
siRNA(Small Interfering RNA)는 다이서를 사용하여 이중가닥 RNA를 길이가 21~23개의 뉴클레오티드 길이로 더 작은 단편으로 절단함으로써 miRNA와 유사한 방식으로 생성되고 기능한다.[9] miRNA와 siRNA는 모두 RISC(RNA-induced silencing complex)를 활성화하여 상보적인 표적 mRNA 서열을 찾고 RNase를 사용하여 RNA를 절단한다.[12] 이것은 차례로 RNA 간섭에 의해 특정 유전자를 침묵시킨다.[13] siRNA와 miRNA는 siRNA가 일반적으로 mRNA 서열에 특이적인 반면 miRNA는 mRNA 서열에 완전히 상보적이지 않다는 점에서 다르다. miRNA는 다른 유전자의 번역을 억제하는 유사한 서열을 가진 표적과 상호작용할 수 있다.[14] 일반적으로 RNA 간섭은 인간과 같은 유기체 내에서 정상적인 과정의 필수적인 부분이며, 암 표적에 대한 진단 및 치료 도구로 연구되고 있는 영역이다.[11]

질병[편집]
황반 변성[편집]
연령 관련 황반변 성은 선진국에서 실명의 주요 원인이다. 이 질병에서 다이서의 역할은 영향을 받은 환자가 망막 색소 상피 (RPE)에서 감소된 수준의 다이서를 나타낸다는 사실이 발견된 후 분명해졌다. RPE에서만 다이서가 결핍된 다이서가 기절된 마우스는 유사한 증상을 나타냈다. 그러나 Drosha 및 Pasha와 같은 중요한 RNAi 경로 단백질이 부족한 다른 마우스는 다이서-knockout 마우스와 같은 황반 변성 증상을 나타내지 않았다. 이 관찰은 RNAi 경로와 무관하고 따라서 si/miRNA 생성의 기능이 아닌 망막 건강에서 다이서의 특정 역할을 제안했다. Alu RNA(alu 요소의 RNA 전사체)라고 하는 RNA의 한 형태는 다이서 수치가 불충분한 환자에서 상승하는 것으로 나타났다. RNA의 이러한 비암호화 가닥은 건강한 망막에서 다이서에 의해 분해될 dsRNA 구조를 형성하는 루프를 형성할 수 있다. 그러나 다이서 수준이 불충분하면 alu RNA가 축적되어 염증의 결과로 RPE가 퇴화된다.[15][16]
암[편집]
악성 암에서 변경된 miRNA 발현 프로파일은 miRNA의 중추적 역할을 제안하고 따라서 암 발병 및 예후에서 다이서(다이서)가 된다. miRNA는 종양 억제인자로 기능할 수 있으므로 변형된 발현은 종양 형성을 유발할 수 있다.[17] 폐암 및 난소암 분석에서 불량한 예후와 감소된 환자 생존 시간은 감소된 다이서 및 드로샤 발현과 상관관계가 있다. 감소된 다이서 mRNA 수준은 진행된 종양 단계와 상관관계가 있다. 그러나 전립선[18] 및 식도와 같은 다른 암에서 높은 다이서 발현은 불량한 환자 예후와 상관관계가 있는 것으로 나타났다. 암 유형 간의 이러한 불일치는 다이서를 포함하는 고유한 RNAi 조절 과정이 종양 유형마다 다르다는 것을 시사한다.[11]
다이서는 DNA 복구에도 관여한다. DNA 손상 복구 및 기타 메커니즘의 효율성 감소의 결과로 감소된 다이서 발현과 함께 포유동물 세포에서 DNA 손상이 증가한다. 예를 들어, 이중가닥 파손(다이서에서 생산)의 siRNA는 이중가닥 파손 복구 메커니즘과 관련된 단백질 복합체에 대한 가이드 역할을 할 수 있으며 염색질 변형을 지시할 수도 있다. 또한, miRNA의 발현 패턴은 이온화 또는 자외선에 의한 DNA 손상의 결과로 변한다. RNAi 메커니즘은 트랜스포존 침묵을 담당하며 다이서가 녹아웃/다운될 때와 같이 RNAi 메커니즘이 없으면 DNA 손상을 일으키는 활성화된 트랜스포존으로 이어질 수 있다. DNA 손상이 축적되면 발암성 돌연변이가 있는 세포가 생겨 종양이 발생할 수 있다.[11]
기타 조건[편집]
신경초종증이 있는 다결절성 갑상선종은 이 유전자의 돌연변이와 관련된 상염색체 우성 상태인 것으로 나타났다.[19]
바이러스 병인[편집]
RNA 바이러스에 의한 감염은 RNAi 캐스케이드를 유발할 수 있다. 식물과 동물 세포를 모두 감염시키는 바이러스에는 RNAi 반응을 억제하도록 설계된 단백질이 포함되어 있기 때문에 다이서가 바이러스 면역에 관여할 가능성이 있다. 인간에서 바이러스 HIV-1, 인플루엔자는 이러한 RNAi 억제 단백질을 인코딩한다. 다이서의 억제는 다이서가 바이러스 dsRNA를 절단하고 생성물을 RISC에 로드하여 바이러스 mRNA의 표적화된 분해를 초래할 수 있기 때문에 바이러스에 유익하다. 따라서 감염과 싸우게 된다. 바이러스 병인의 또 다른 잠재적 메커니즘은 세포 miRNA 경로를 억제하는 방법으로 다이서를 차단하는 것이다.[20]
곤충[편집]
곤충은 다이서를 강력한 항바이러스 제로 사용할 수 있다. 이 발견은 모기가 잠재적으로 치명적인 arboviruses (웨스트 나일 바이러스, 뎅기열 및 황열병)를 포함한 많은 바이러스성 질병의 전염에 책임이 있다는 점을 고려할 때 특히 중요하다.[21] 모기(Aedes aegypti )는 이러한 바이러스의 매개체 역할을 하지만 바이러스의 의도된 숙주는 아니다. 감염은 암컷 모기가 알을 낳기 위해 척추동물의 혈액이 필요하기 때문에 발생한다. 곤충의 RNAi 경로는 다른 동물의 경로와 매우 유사하다. 다이서-2는 바이러스 RNA를 절단하여 한 가닥이 RNAi 제품 생산을 위한 템플릿 역할을 하고 다른 가닥이 분해되는 RISC 복합체에 로드한다. RNAi 경로의 비기능적 구성요소로 이어지는 돌연변이를 가진 곤충은 그들이 가지고 있는 바이러스에 대한 증가된 바이러스 부하를 나타내거나 그들이 숙주인 바이러스에 대한 증가된 감수성을 보여준다. 인간과 유사하게 곤충 바이러스는 RNAi 경로를 피하기 위한 메커니즘을 진화했다. 예를 들어, 초파리 C 바이러스는 dsRNA에 결합하여 다이서 절단 및 RISC 로딩으로부터 보호하는 단백질 1A를 암호화한다. Heliothis virescens ascovirus 3a는 dsRNA 기질과 경쟁할 수 있고 RISC 로딩을 방지하기 위해 siRNA 이중체를 분해할 수 있는 다이서의 RNase III 그룹과 유사한 RNase III 그룹 효소를 인코딩한다.[22]
진단 및 치료 애플리케이션[편집]
다이서는 효소의 발현 수준에 따라 종양이 체내에 존재하는지 여부를 식별하는 데 사용할 수 있다. 연구에 따르면 암에 걸린 많은 환자들이 다이서의 발현 수준이 감소했다. 동일한 연구에서 낮은 다이서 발현은 낮은 환자 생존 기간과 상관관계가 있음을 보여주었다.[11] 다이서는 진단 도구와 함께 외래 siRNA를 정맥 주사하여 유전자 침묵을 유발하여 환자를 치료하는 데 사용할 수 있다.[23]
siRNA는 마우스와 같은 포유류 종에서 두 가지 방식으로 전달되는 것으로 나타났다. 한 가지 방법은 다이서 기능이 필요하지 않은 시스템에 직접 주입하는 것이다. 또 다른 방법은 다이서에 의해 siRNA로 절단되는 짧은 헤어핀 RNA를 인코딩하는 플라스미드에 의해 도입하는 것이다.[24]
다이서를 사용하여 siRNA를 치료적으로 생산하는 이점 중 하나는 항체 또는 소분자 억제제와 같이 현재 사용되고 있는 것과 비교하여 영향을 미칠 수 있는 표적의 특이성과 다양성이다. 일반적으로 저분자 억제제는 견디기 힘든 부작용과 함께 특이성 면에서 어렵다. 항체는 siRNA만큼 특이적이지만 리간드 나 표면 수용체에 대해서만 사용할 수 있다는 한계가 있다. 한편, 세포내 흡수 효율이 낮다는 것이 siRNA 주입의 주요 걸림돌이다.[11] 주입된 SiRNA는 혈액 내 안정성이 좋지 않아 비특이적 면역 자극을 유발한다.[25] 또한, miRNA가 mRNA에 부착되기 위해서는 6-8개의 뉴클레오티드 염기쌍만 필요하기 때문에 치료학적으로 miRNA를 생산하는 것은 특이성이 부족하다.[26]
다이서 유사 단백질[편집]
식물 게놈은 동물 및 곤충 다이서와 유사한 기능 및 단백질 도메인을 갖는 다이서 유사 단백질을 암호화한다. 예를 들어, 모델 유기체 Arabidopsis thaliana에서 4개의 다이서 유사 단백질이 만들어지고 DCL1에서 DCL4로 지정된다. DCL1은 역반복체로부터 miRNA 생성 및 sRNA 생성에 관여한다. DCL2는 바이러스 면역 및 방어를 돕는 시스 작용 안티센스 전사체로부터 siRNA를 생성한다. DCL3은 염색질 수정을 돕는 siRNA를 생성하고 DCL4는 전사 후 수준에서 트랜스-작용 siRNA 대사 및 전사 침묵에 관여한다. 또한 DCL 1과 3은 Arabidopsis 개화에 중요하다. Arabidopsis에서 DCL 녹아웃은 심각한 발달 문제를 일으키지 않다.
다이서 메커니즘이 많은 유기체의 일반적인 방어 전략이기 때문에 쌀과 포도도 DCL을 생성한다. 벼는 생산하는 5개의 DCL에 대해 다른 기능을 진화시켰으며 기능과 발달에서 애기장대보다 더 중요한 역할을 한다. 또한, 발현 패턴은 쌀의 다른 식물 세포 유형 간에 다르지만 Arabidopsis에서의 발현은 더 균질하다. 벼 DCL 발현은 가뭄, 염분 및 추위를 포함한 생물학적 스트레스 조건에 의해 영향을 받을 수 있으므로 이러한 스트레스 요인은 식물의 바이러스 내성을 감소시킬 수 있다. Arabidopsis와 달리 DCL 단백질의 기능 상실은 벼의 발달 결함을 유발한다.[27]
같이 보기[편집]
각주[편집]
- ↑ 가 나 “[네이버 지식백과] 다이서 [Dicer] (분자·세포생물학백과)”. 2022년 2월 26일에 확인함.
- ↑ “Role of Dicer in posttranscriptional RNA silencing”. 《Current Topics in Microbiology and Immunology》 320: 77–97. 2008. doi:10.1007/978-3-540-75157-1_4. ISBN 978-3-540-75156-4. PMID 18268840.
- ↑ “Transcriptional control of gene expression by microRNAs”. 《Cell》 140 (1): 111–22. Jan 2010. doi:10.1016/j.cell.2009.12.023. PMID 20085706.
- ↑ 가 나 “Structure of the human Dicer-TRBP complex by electron microscopy”. 《Structure》 17 (10): 1326–32. Oct 2009. doi:10.1016/j.str.2009.08.013. PMC 2880462. PMID 19836333.
- ↑ 가 나 “Structural basis for double-stranded RNA processing by Dicer”. 《Science》 311 (5758): 195–8. Jan 2006. Bibcode:2006Sci...311..195M. doi:10.1126/science.1121638. PMID 16410517.
- ↑ “Entrez Gene: DICER1 Dicer1, Dcr-1 homolog (Drosophila)”.
- ↑ “Molecular cloning and characterization of a novel human gene (HERNA) which encodes a putative RNA-helicase”. 《Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression》 1490 (1–2): 163–9. Jan 2000. doi:10.1016/S0167-4781(99)00221-3. PMID 10786632.
- ↑ “Dicing and slicing: the core machinery of the RNA interference pathway”. 《FEBS Letters》 579 (26): 5822–9. Oct 2005. doi:10.1016/j.febslet.2005.08.079. PMID 16214139.
- ↑ 가 나 다 “Phosphate and R2D2 restrict the substrate specificity of Dicer-2, an ATP-driven ribonuclease”. 《Molecular Cell》 42 (2): 172–84. Apr 2011. doi:10.1016/j.molcel.2011.03.002. PMC 3115569. PMID 21419681.
- ↑ “An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells”. 《Nature》 404 (6775): 293–6. Mar 2000. Bibcode:2000Natur.404..293H. doi:10.1038/35005107. PMID 10749213.
- ↑ 가 나 다 라 마 바 “The dicey role of Dicer: implications for RNAi therapy”. 《Cancer Research》 70 (7): 2571–4. Apr 2010. doi:10.1158/0008-5472.CAN-09-2536. PMC 3170915. PMID 20179193.
- ↑ “The contributions of dsRNA structure to Dicer specificity and efficiency”. 《RNA》 11 (5): 674–82. May 2005. doi:10.1261/rna.7272305. PMC 1370754. PMID 15811921.
- ↑ Watson JD (2008). 《Molecular Biology of the Gene》. San Francisco, CA: Cold Spring Harbor Laboratory Press. 641–648쪽. ISBN 978-0-8053-9592-1.
- ↑ “MicroRNAs and small interfering RNAs can inhibit mRNA expression by similar mechanisms”. 《Proceedings of the National Academy of Sciences of the United States of America》 100 (17): 9779–84. Aug 2003. Bibcode:2003PNAS..100.9779Z. doi:10.1073/pnas.1630797100. PMC 187842. PMID 12902540.
- ↑ “Vision: Dicer leaps into view”. 《Nature》 471 (7338): 308–9. Mar 2011. Bibcode:2011Natur.471..308M. doi:10.1038/471308a. PMID 21412326.
- ↑ “DICER1 loss and Alu RNA induce age-related macular degeneration via the NLRP3 inflammasome and MyD88”. 《Cell》 149 (4): 847–59. May 2012. doi:10.1016/j.cell.2012.03.036. PMC 3351582. PMID 22541070.
- ↑ “The role of dicer in DNA damage repair”. 《International Journal of Molecular Sciences》 13 (12): 16769–78. 2012. doi:10.3390/ijms131216769. PMC 3546719. PMID 23222681.
- ↑ “Up-regulation of dicer, a component of the MicroRNA machinery, in prostate adenocarcinoma”. 《The American Journal of Pathology》 169 (5): 1812–20. Nov 2006. doi:10.2353/ajpath.2006.060480. PMC 1780192. PMID 17071602.
- ↑ Rivera B, Nadaf J, Fahiminiya S, Apellaniz-Ruiz M, Saskin A, Chong AS, Sharma S, Wagener R, Revil T, Condello V, Harra Z, Hamel N, Sabbaghian N, Muchantef K, Thomas C, de Kock L, Hébert-Blouin MN, Bassenden AV, Rabenstein H, Mete O, Paschke R, Pusztaszeri MP, Paulus W, Berghuis A, Ragoussis J, Nikiforov YE, Siebert R, Albrecht S, Turcotte R, Hasselblatt M, Fabian MR, Foulkes WD (2019) DGCR8 microprocessor defect characterizes familial multinodular goiter with schwannomatosis.
- ↑ “The interplay between virus infection and the cellular RNA interference machinery”. 《FEBS Letters》 580 (12): 2896–902. May 2006. doi:10.1016/j.febslet.2006.02.070. PMC 7094296. PMID 16563388.
- ↑ “Mosquito-borne Diseases”. National Center for Infections Disease, Center for Disease Control and Prevention. 2014년 1월 31일에 원본 문서에서 보존된 문서. 2014년 4월 22일에 확인함.
- ↑ “The long and short of antiviral defense: small RNA-based immunity in insects”. 《Current Opinion in Virology》 7: 19–28. Aug 2014. doi:10.1016/j.coviro.2014.03.010. PMID 24732439.
- ↑ “Intravenous injection of siRNA directed against hypoxia-inducible factors prolongs survival in a Lewis lung carcinoma cancer model”. 《Cancer Gene Therapy》 16 (3): 195–205. Mar 2009. doi:10.1038/cgt.2008.71. PMID 18818708.
- ↑ “Gene silencing by RNA interference is being used routinely to study gene function in cultured mammalian cells”. 《Life Technologies》. 2014년 10월 20일에 원본 문서에서 보존된 문서. 2014년 4월 23일에 확인함.
- ↑ “Cancer siRNA therapy by tumor selective delivery with ligand-targeted sterically stabilized nanoparticle”. 《Nucleic Acids Research》 32 (19): e149. 2004. doi:10.1093/nar/gnh140. PMC 528817. PMID 15520458.
- ↑ “Argonaute HITS-CLIP decodes microRNA-mRNA interaction maps”. 《Nature》 460 (7254): 479–86. Jul 2009. Bibcode:2009Natur.460..479C. doi:10.1038/nature08170. PMC 2733940. PMID 19536157.
- ↑ “Dicer-like (DCL) proteins in plants”. 《Functional & Integrative Genomics》 9 (3): 277–86. Aug 2009. doi:10.1007/s10142-009-0111-5. PMID 19221817.
