단세포 분석: 두 판 사이의 차이
"Single-cell analysis" 문서를 번역하여 만듦 |
(차이 없음)
|
2022년 4월 27일 (수) 23:29 판
![]() 작업 중
작업 중
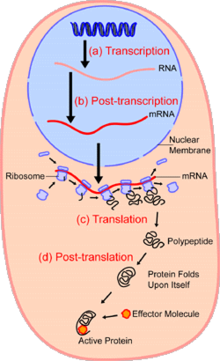
세포생물학에서, 단일 세포 분석은 단일 세포 수준에서 유전체학, 전사체학, 단백질체학, 대사체학 및 세포 간 상호작용 에 대한 연구이다. [1] [2] [3] 진핵 및 원핵 세포군 모두에서 이질성(heterogeneity)이 보여지므로, 단일 세포를 분석하면 대량의 세포 집단을 연구할 때 볼 수 없는 메커니즘을 발견할 수 있다. [4] FACS( fluorescence-activated cell sorting )와 같은 기술을 사용하면 시료에서 특정 단일 세포를 정확하게 분리할 수 있다. 이는 처리량이 많은 단일 세포 분할 기술[5] [6] [7]을 통해 동시에 수백 또는 수천 개의 단일 세포의 분자 분석이 가능하다; 이는 특히 유전자형이 동일한 세포에서 전사체 변이를 분석하는 데 유용하며, 그 외 감지되지 않는 세포 하위 유형을 정의할 수 있게 된다. 새로운 기술의 개발은 단일 세포의 게놈과 전사체를 분석하고 [8] 단백질체와 대사체 를 정량하는 능력을 향상 시키고 있다. [9] [10] [11] 질량 분석 기술은 단일 세포의 단백질체 및 대사체 분석을 위한 중요한 분석 기법이 되었다. [12] [13] 최근 발전으로 수백 개의 단일 세포에서 수천 개의 단백질 정량이 가능하게 되었고 [14], 이로 인해 새로운 유형의 분석이 가능해졌다. [15] [16] In situ sequencing 및 형광 원위치 혼성화 (Fluorescence in situ hybridzation, FISH)는 세포를 분리하지 않아도 되며 조직 분석에 점점 더 많이 사용되고 있다. [17]
단일 세포 분리
많은 단일 세포 분석 기술은 각 세포의 분리를 필요로 한다. 현재 단일 세포 분리에 사용되는 방법으로 : 유전영동 디지털 분류(dielectrophoretic digital sorting), FACS, 동수집유장(hydrodynamic traps), 레이저 포착 현미해부(laser capture microdissection), 미세 유체 공학(microfluidics), 현미조작(micromanipulation), 연속희석법(serial dilution) 등이 있다.
관련 기술
유전영동 디지털 분류법(dieletrophoretic digital sorting method)은 미세유체칩의 반도체 제어 전극을 활용하여 유전영동(DEP) 케이지에서 단일 세포를 포획하는 기법이다. 세포의 식별은 형광 마커의 이미지 관찰을 통해 검증될 수 있다. 또한. 또한, 유세포에서 DEP 케이지의 반도체 제어 동작을 통해 정확히 전달되었는지 확인할 수 있다.
유체역학적 포획은 수동적 미세유체 수송에 의해 각각 주어진 시간에 "trap"에 각 세포를 분리할 수 있다. 분리된 세포의 수는 시스템의 trap 수에 기반하여 조작할 수 있다.
레이저 포착 현미해부(Laser Capture Microdissection) 기술은 보고자 하는 조직 샘플에서 개체 세포 또는 특정 부분을 해부하고 분리하기 위해 레이저를 사용한다. 이 방법은 현미경으로 세포를 관찰하여 분석할 부분을 식별하고 표지하여 레이저로 세포를 절단하게 된다. 그런 다음 분석을 위해 세포를 추출한다.
수동 단일 세포 선택(manual single cell picking)은 세포를 현미경으로 관찰하고 마이크로피펫 을 사용하여 선택하는 방법이다. [18] [19]
미세 유체 공학(microfluidics)는 추후 분석을 위힌 개별 세포의 분리에 사용된다. 다음 원칙은 단일 세포의 분리를 위한 다양한 미세 유체 과정을 간략하게 설명하고 있다: droplet-in-oil 기반 분리, 공기막 밸브(pneumatic membrane valving) 및 유체 역학 세포 트랩(hydrodynamic cell traps). Droplet-in-oil 기반 미세 유체는 분리된 aqueous droplet을 유지하기 위해 오일로 채워진 채널을 사용한다. 이를 통해서 단일 세포는 오일 기반 채널 내부에 들어있으며 분리될 수 있다. 공압 멤브레인 밸브는 공기압 조작을 사용하여 멤브레인 편향에 의해 개별 세포를 분리합니다. 압력의 조절로 미세 유체 네트워크에서 채널을 열거나 닫을 수 있다. 일반적으로 시스템 운영자가 필요하며 제한된 처리량을 가지고 있다.
라만 핀셋(Ramen tweezers) 기술은 라만 분광법과 광학 핀셋이 결합된 기법으로 레이저 빔을 사용하여 세포를 포획하고 조작하는 데 사용된다. [20]
유체역학 기반 미세유체 바이오칩의 개발은 수년에 걸쳐 증가하고 있습니다. 이 기술에서 세포는 단일 세포 분석(SCA)을 위해 특정 영역에 갇혀 있습니다. 이것은 일반적으로 광학, 전기, 자기 또는 음향과 같은 외부 힘 필드의 적용 없이 발생합니다. 세포의 자연 상태에서 SCA에 대한 통찰력을 탐구할 필요가 있으며 이러한 기술의 개발은 해당 연구에 매우 중요합니다. 연구원들은 시장 및 연구원 요구에 맞는 바이오칩 장치를 개발할 필요성을 강조했습니다. 유체역학적 미세유체학은 패시브 랩온칩 애플리케이션의 개발을 용이하게 합니다.
단일 세포 대사체학의 목적은 암, 줄기 세포, 노화 및 약물 내성 발달과 같은 주요 생물학적 주제의 분자 수준에서 더 나은 이해를 얻는 것입니다. 일반적으로 대사체학의 초점은 대부분 세포가 분자 수준에서 환경 스트레스를 처리하는 방법을 이해하고 세포 기능에 대한 보다 역동적인 이해를 제공하는 것입니다. [21]
세포 간 상호작용
세포 간 상호작용은 안정적이고 일시적인 상호작용이다.
참고문헌
- ↑ “Single cell analysis: the new frontier in 'omics'”. 《Trends in Biotechnology》 28 (6): 281–90. June 2010. doi:10.1016/j.tibtech.2010.03.002. PMC 2876223. PMID 20434785.
- ↑ “Computation and measurement of cell decision making errors using single cell data”. 《PLOS Computational Biology》 13 (4): e1005436. April 2017. Bibcode:2017PLSCB..13E5436H. doi:10.1371/journal.pcbi.1005436. PMC 5397092. PMID 28379950.
- ↑ “Automated profiling of individual cell-cell interactions from high-throughput time-lapse imaging microscopy in nanowell grids (TIMING)”. 《Bioinformatics》 31 (19): 3189–97. October 2015. doi:10.1093/bioinformatics/btv355. PMC 4693004. PMID 26059718.
- ↑ “Cellular heterogeneity: do differences make a difference?”. 《Cell》 141 (4): 559–63. May 2010. doi:10.1016/j.cell.2010.04.033. PMC 2918286. PMID 20478246.
- ↑ “Single Cell Isolation and Analysis”. 《Frontiers in Cell and Developmental Biology》 4: 116. 2016년 10월 25일. doi:10.3389/fcell.2016.00116. PMC 5078503. PMID 27826548.
- ↑ “Miniaturization Technologies for Efficient Single-Cell Library Preparation for Next-Generation Sequencing”. 《Journal of Laboratory Automation》 21 (4): 557–67. August 2016. doi:10.1177/2211068216630741. PMC 4948133. PMID 26891732.
- ↑ “Massively parallel digital transcriptional profiling of single cells”. 《Nature Communications》 8: 14049. January 2017. Bibcode:2017NatCo...814049Z. doi:10.1038/ncomms14049. PMC 5241818. PMID 28091601.
- ↑ “Single-Cell Gene Network Analysis and Transcriptional Landscape of MYCN-Amplified Neuroblastoma Cell Lines”. 《Biomolecules》 11 (2): 177. January 2021. doi:10.3390/biom11020177. PMC 7912277. PMID 33525507.
- ↑ “Single-Cell Whole-Genome Amplification and Sequencing: Methodology and Applications”. 《Annual Review of Genomics and Human Genetics》 16 (1): 79–102. 2015. doi:10.1146/annurev-genom-090413-025352. PMID 26077818.
- ↑ “Single-Cell Transcriptional Analysis”. 《Annual Review of Analytical Chemistry》 10 (1): 439–462. June 2017. doi:10.1146/annurev-anchem-061516-045228. PMID 28301747.
- ↑ “A new toolbox for assessing single cells”. 《Annual Review of Chemical and Biomolecular Engineering》 5: 455–77. 2014. doi:10.1146/annurev-chembioeng-060713-035958. PMC 4309009. PMID 24910919.
- ↑ “Categorizing Cells on the Basis of their Chemical Profiles: Progress in Single-Cell Mass Spectrometry”. 《Journal of the American Chemical Society》 139 (11): 3920–3929. March 2017. doi:10.1021/jacs.6b12822. PMC 5364434. PMID 28135079.
- ↑ “Single-Cell Mass Spectrometry Approaches to Explore Cellular Heterogeneity”. 《Angewandte Chemie》 57 (17): 4466–4477. April 2018. doi:10.1002/anie.201709719. PMID 29218763.
- ↑ “Single-cell protein analysis by mass spectrometry”. 《Current Opinion in Chemical Biology》 60: 1–9. June 2020. arXiv:2004.02069. doi:10.1016/j.cbpa.2020.04.018. ISSN 1367-5931. PMC 7767890. PMID 32599342.
- ↑ “Transformative Opportunities for Single-Cell Proteomics”. 《Journal of Proteome Research》 17 (8): 2565–2571. August 2018. doi:10.1021/acs.jproteome.8b00257. PMC 6089608. PMID 29945450.
- ↑ “Unpicking the proteome in single cells”. 《Science》 367 (6477): 512–513. January 2020. Bibcode:2020Sci...367..512S. doi:10.1126/science.aaz6695. PMC 7029782. PMID 32001644.
- ↑ “De Novo Gene Expression Reconstruction in Space”. 《Trends in Molecular Medicine》 23 (7): 583–593. July 2017. doi:10.1016/j.molmed.2017.05.004. PMC 5514424. PMID 28571832.
- ↑ “Technologies for Single-Cell Isolation”. 《International Journal of Molecular Sciences》 16 (8): 16897–919. July 2015. doi:10.3390/ijms160816897. PMC 4581176. PMID 26213926.
- ↑ “Energy Charge, Redox State, and Metabolite Turnover in Single Human Hepatocytes Revealed by Capillary Microsampling Mass Spectrometry”. 《Analytical Chemistry》 87 (20): 10397–405. October 2015. doi:10.1021/acs.analchem.5b02502. PMID 26398405.
- ↑ 〈Analysis of single eukaryotic cells using Raman Tweezers〉. 《Single-Cell Analysis》. Methods in Molecular Biology 853. Humana Press. January 2012. 151–67쪽. doi:10.1007/978-1-61779-567-1_12. ISBN 978-1-61779-566-4. PMID 22323146.
- ↑ “Single-cell metabolomics: analytical and biological perspectives”. 《Science》 342 (6163): 1243259. December 2013. doi:10.1126/science.1243259. PMID 24311695.
추가 읽기
- Lim, S. B., Lim, C. T., & Lim, W. T. (2019). Single-cell analysis of circulating tumor cells: why heterogeneity matters. Cancers, 11(10), 1595. PMID 31635038PMID 31635038 PMC 6826423 doi 10.3390/cancers11101595
- Zhou, W. M., Yan, Y. Y., Guo, Q. R., Ji, H., Wang, H., Xu, T. T., ... & Zhang, J. Y. (2021). Microfluidics applications for high-throughput single cell sequencing. Journal of Nanobiotechnology, 19(1), 1-21. PMID 34635104PMID 34635104 PMC 8507141 doi 10.1186/s12951-021-01045-6
- Ding, L., Radfar, P., Rezaei, M., & Warkiani, M. E. (2021). An easy-to-operate method for single-cell isolation and retrieval using a microfluidic static droplet array. Microchimica Acta, 188(8), 242. PMID 34226955PMID 34226955 doi 10.1007/s00604-021-04897-9
- Luo, C., Liu, H., Xie, F., Armand, E. J., Siletti, K., Bakken, T. E., ... & Ecker, J. R. (2022). Single nucleus multi-omics identifies human cortical cell regulatory genome diversity. Cell Genomics, 2(3), 100107. https://doi.org/10.1016/j.xgen.2022.100107 Single-cell multi-omic method, simultaneously profiling transcriptome, DNA methylome, and chromatin accessibility
- Descamps, L., Le Roy, D., & Deman, A. L. (2022). Microfluidic-Based Technologies for CTC Isolation: A Review of 10 Years of Intense Efforts towards Liquid Biopsy. International Journal of Molecular Sciences, 23(4), 1981. PMID 35216097PMID 35216097 PMC 8875744 doi 10.3390/ijms23041981
